008-综述之单细胞转录组分析最佳思路
刘小泽写于2020.5.6-5.8
历时三天,终于理解完!不仅仅是综述,更不是纯翻译文,而是将其中重要的知识点和之前个人所学结合起来 ”一阳指和狮吼功合并为一整招“
1 文章信息
题目:Current best practices in single-cell RNA-seq analysis: a tutorial
发表日期:2019年6月19日
杂志:Mol Syst Biol
文章在:https://www.embopress.org/doi/10.15252/msb.20188746
DOI:https://doi.org/10.15252/msb.20188746

2 摘要
单细胞领域日新月异,大量的工具被开发出来,但很难去判断是否好用,而且如何组建一个分析流程是一个难点。本文将详细介绍单细胞转录组数据分析的步骤,包括预处理(质控、归一化标准化、数据矫正、挑选基因、降维)以及细胞和基因层面的下游分析。并且作者将整个流程应用在了一个公共数据集作为展示(详细说明在:https://www.github.com/theislab/single-cell-tutorial),目的是帮助新入坑用户建立一个知识体系,已入坑用户更新知识体系。
3 前言
需要注意,虽然在原文链接中这些文献链接可以打开,但会链接到原文的该文献位置,而不是直接打开该文献
现在已经可以利用scRNA研究斑马鱼、青蛙、涡虫的细胞异质性(Briggs et al, 2018; Plass et al, 2018; Wagner et al, 2018) ,重新理解以前的细胞群体,但这个领域面临的一个问题就是没有成熟的标准化流程。标准化之路的困难有:大量分析方法和工具的诞生(截止2019.3.7 已经有385种工具)、爆炸式增长的数据量(Angerer et al, 2017; Zappia et al, 2018)。另外根据不同研究目的,各种分支也突显,例如在细胞分化过程中预测细胞命运(La Manno et al, 2018)。在我们眼界大开的同时,分析流程标准化就变得更加困难。
在未来分析流程标准化之路上,困难还会存在于技术整合层面。比如现在大量的scRNA工具都是用R和Python写的,跨平台分析需求在增长,而对编程语言的喜好也决定了工具的选择。很多好用的分析工具将自己限制在用各自的编程语言开发的环境中,例如Seurat、Scater、Scanpy。
接下来,就一起看看作者列出了哪些他认为比较好的软件和流程吧
先上一个scRNA分析总体流程图:
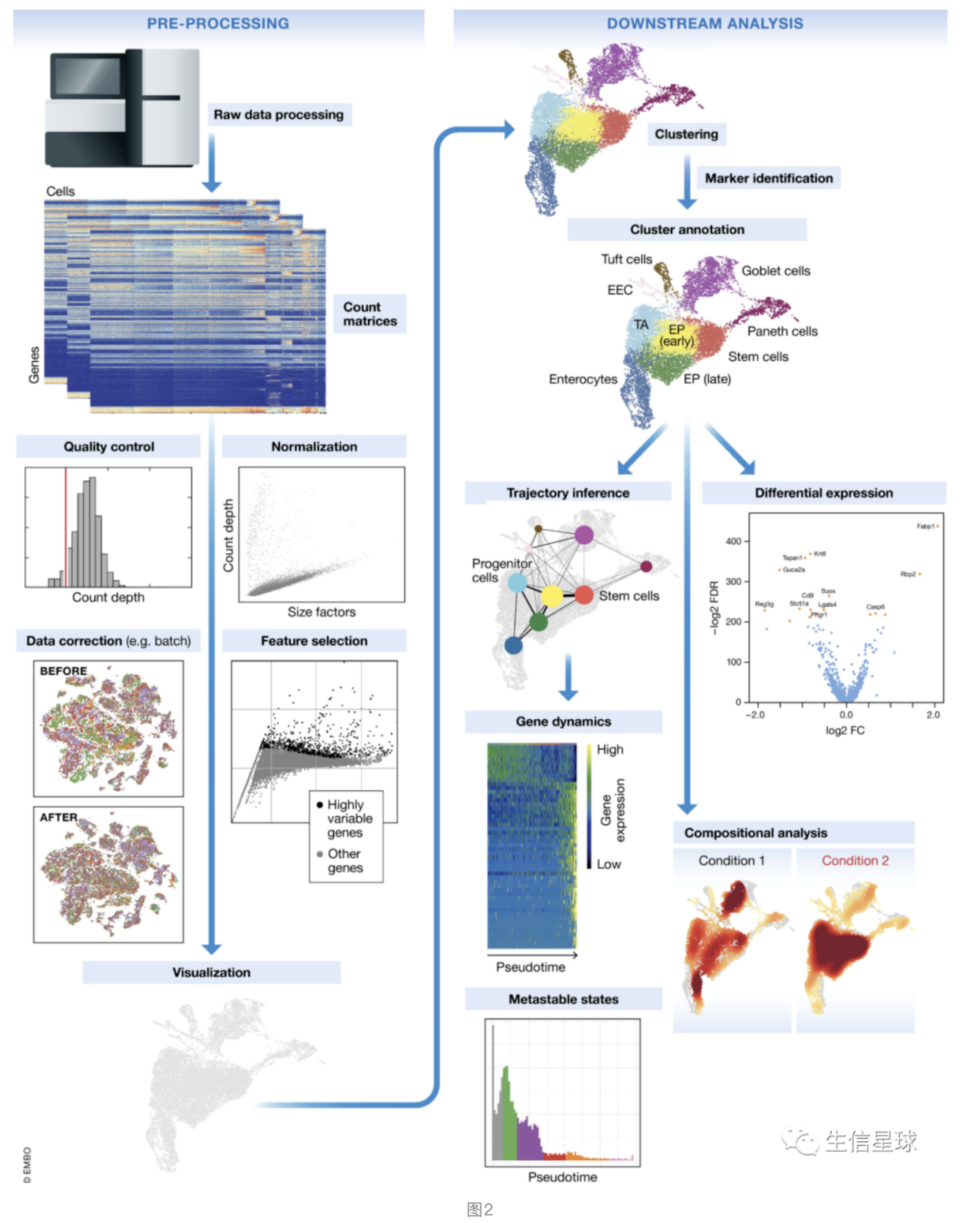
4 预处理和可视化
4.1 首先看一下实验过程
比较详细的介绍可以看:Ziegenhain et al ( 2017); Macosko et al ( 2015); Svensson et al ( 2017).
原文描述的关键点是:
4步走:Typical workflows incorporate single‐cell dissociation, single‐cell isolation, library construction, and sequencing. 组织裂解=》细胞分离=》文库构建=》测序
第一步:As a first step, a single‐cell suspension is generated in a process called single‐cell dissociation in which the tissue is digested.
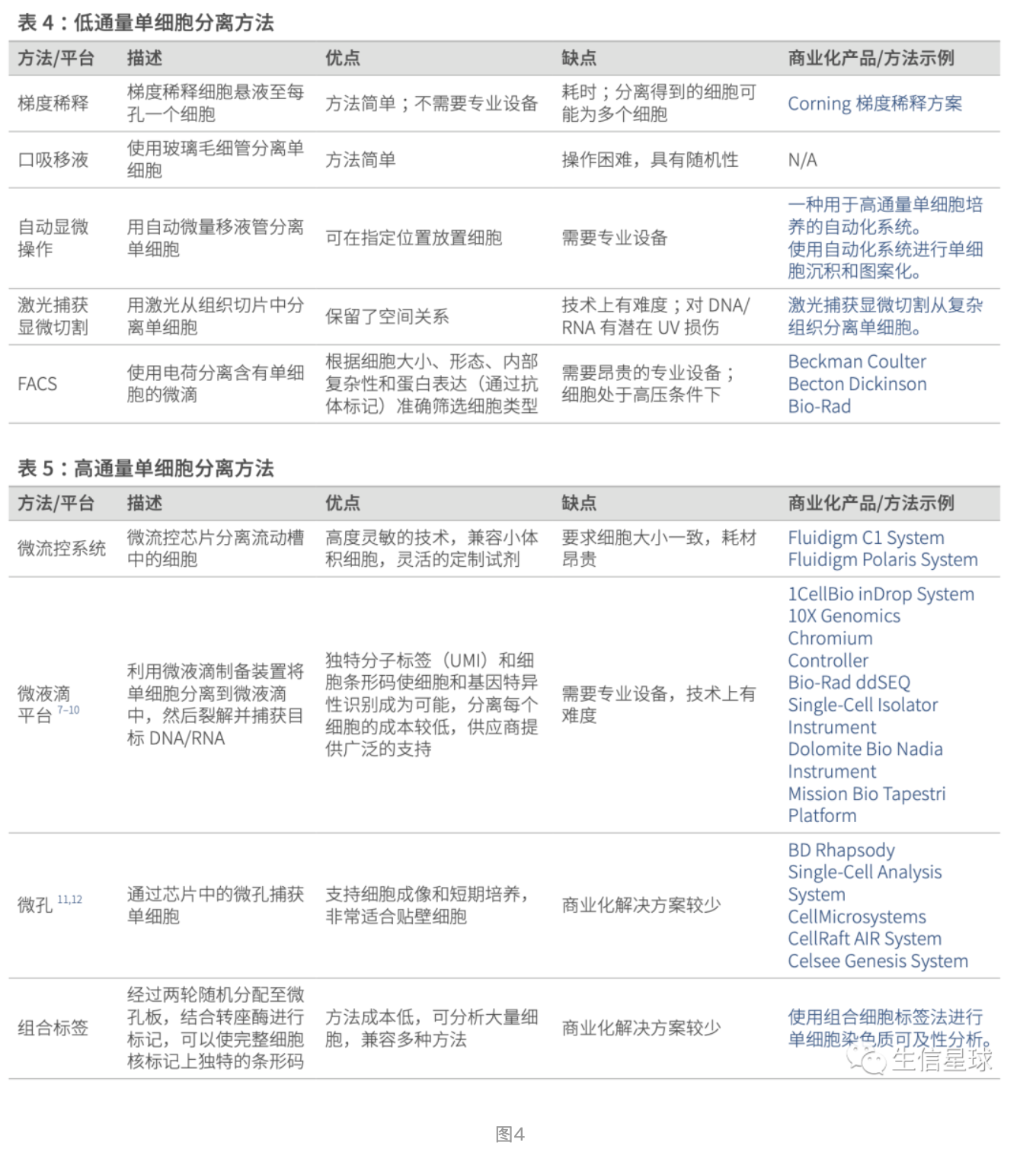
第二步:To profile the mRNA in each cell separately, cells must be isolated. 写了主要的2种方法:plate‐based、droplet‐based,当然也都存在一些问题: In both cases, errors can occur that lead to multiple cells being captured together (doublets or multiplets), non‐viable cells being captured, or no cell being captured at all (empty droplets/wells)
第三步:Each well or droplet contains the necessary chemicals to break down the cell membranes and perform library construction. Furthermore, many experimental protocols also label captured molecules with a unique molecular identifier (UMI).
UMI的作用主要是区分:UMIs allow us to distinguish between amplified copies of the same mRNA molecule and reads from separate mRNA molecules transcribed from the same gene.
第四步:Libraries are labelled with cellular barcodes and pooled together (multiplexed) for sequencing.
感觉原文描述的还没有illumina给出的详细,那么就看看illumina的图文并茂版:
illumina单细胞测序工作流程:关键步骤和注意事项:http://web.illumina.com.cn/landing/products_view.asp?newsid=324
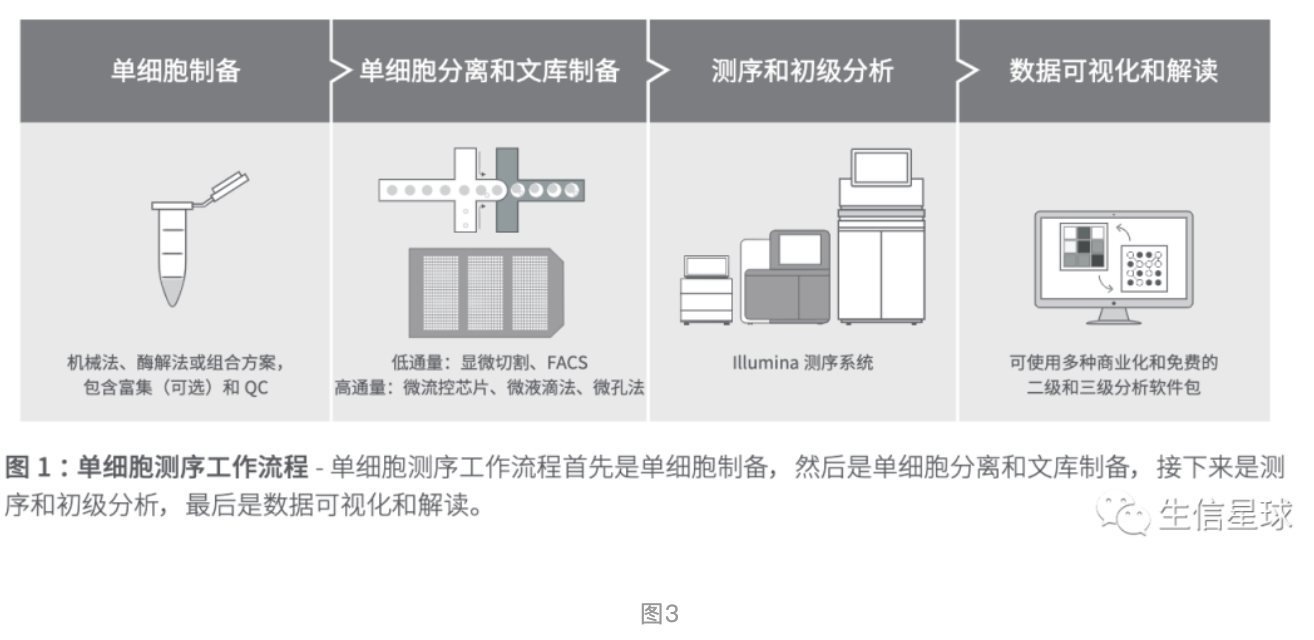
原始测序数据要经过处理得到表达矩阵,注意这里有两种表述方式:molecular counts (count matrices) 【也即是使用UMI的】和 read counts (read matrices),取决于是否使用UMI。而作者介绍的流程中,默认使用 count matrices,除非readmatrices和 count matrices得到的结果存在差异,才会特别介绍read matrices
关于比较read and molecule counts,有人写了一个R流程:https://jdblischak.github.io/singleCellSeq/analysis/compare-reads-v-molecules.html
原始数据处理工具主要有:CellRanger、indrops、SEQC、zUMIs
它们主要做了这么几件事:
- read quality control (QC)
- assigning reads to their cellular barcodes and mRNA molecules of origin (also called “demultiplexing”)
- genome alignment
- quantification
得到的矩阵行是转录本,列是barcodes【这里用barcodes而不是直接叫细胞,是因为不同细胞的reads也可能属于同一个barcode =》如果出现一孔/液滴多细胞(doublet情况),那么barcode在多个细胞都是一样的】当然也会出现有barcode但实际没有细胞的情况(一个孔/液滴没有细胞即droplet,但这个孔/液滴也会赋予barcode)
反正记住:barcode和孔/液滴是对应的,但一个孔/液滴中有一个细胞还是多个细胞或者没有细胞,都会存在barcode。只不过最后可能会看到:多个细胞对应一个barcode、即使没有细胞也会有barcode这样的情况
关于10X实验环节,可以看我之前写的:https://mp.weixin.qq.com/s/0DEybX7GnuDFhfY1uj9t9A

4.2 质控
在正式分析之前,先要确定barcode是不是对应真正的细胞(上面已经了解了barcode和细胞的关系),也就是进行Cell QC,主要考虑三个因素(这几个因素也就是现在流程中常用的过滤指标):
- the number of counts per barcode (count depth)
- the number of genes per barcode
- the fraction of counts from mitochondrial genes per barcode
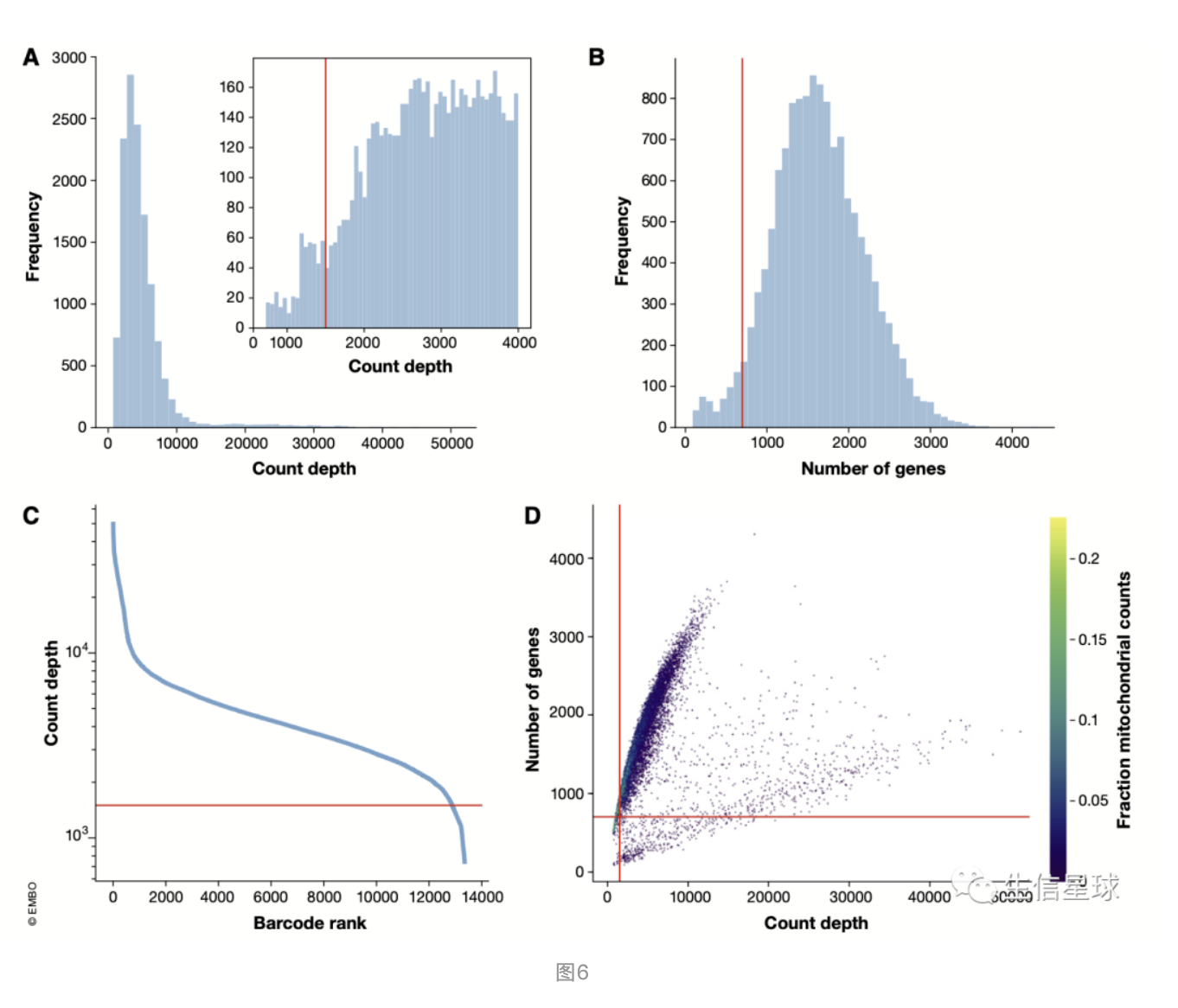
从下面👇图中,感觉过滤的一个方向就是:保留大山头,去掉小山头(把略有增长但不碍大局的小山头炸掉)
先看图A:其中这个小的直方图就是把count depth小于4000的放大,这里设定了一个阈值1500,也就是一个barcode中至少有1500的表达量
图B:每个细胞中包含的基因数直方图。可以看到横坐标有一个小的峰在400附近,这里设定的阈值是700
图C:依旧是看count depth。从高到低排列count depth值,可以过滤一些空的液滴(empty droplets),看到从”肘部“也就是纵坐标1500左右开始迅速下降
图D:看线粒体比例。如果占比很高并且细胞类型不是线粒体特别丰富的那种(如心肌细胞),可能说明这个细胞本身的基因数不多并且总体表达量也不高
以上三个指标固然重要,但如果只关注其中某一个,也会产生误导作用,所以作者建议看问题一定要全面,并且要把数据和生物学知识结合起来。作者举了个例子:比如线粒体表达量相对较高的细胞也可能参与了呼吸过程。细胞总体表达量低或者基因数量少,也可能是因为当时取的细胞处于静止;细胞表达量很高,也可能因为本身细胞体积就比较大。的确,细胞与细胞之间的总表达量还是存在较大差异的。未来也许QC会提供更多的选择。
除了检查细胞完整度,QC还要进行转录本层面上的检查。原始的count矩阵一般包含超过20000个基因。这里一般要根据在细胞中有表达的数量进行过滤,但这个阈值要根据总体细胞数和预计的分群情况来灵活调整。比如有的细胞类型本身就数量比较少(也许就50个),那么如果我们要设定”在少于50个细胞中有表达的基因“这种条件,那么可能会丢失那些总共就50个细胞中的marker基因,最终导致鉴定的细胞亚群会缺失。
质控的目的就是给下游提供更高质量的数据,但一开始谁也不知道这个质量高不高,只能先进行下游分析,看看结果(比如细胞分群结果)再判断。尤其是针对异质性高的细胞群体
文章又额外介绍了一些QC指标: https://www.embopress.org/action/downloadSupplement?doi=10.15252%2Fmsb.20188746&file=msb188746-sup-0001-Appendix.pdf 比如在CellRanger的结果报告中就会有:Q30指标(一般要高于60-70%)、比对到外显子的比例(一般认为比对到非外显子区域超过40%就造成了测序的浪费)、看看实验中用了多少个细胞,以及结果表达矩阵得到多个barcode
小结
- 三种QC指标(the number of genes、the count depth 、the fraction of mitochondrial reads)要放在一起思考,而不是单独看某一个
- 先尽可能地设定宽泛的QC阈值,如果下游聚类无法解释再回过头来反思QC
- 如果看到每个样本的QC指标分布不同,那么就要对每个样本分别设定阈值,而不是一刀切
4.3 归一化/标准化
**作为背景知识,首先来看:**归一化和标准化的区别(https://cloud.tencent.com/developer/article/1486102)但二者的界限也没有特别明显,也没有必要把这两个概念分的特别清楚。只要清楚它们大概的使用范围就可以了:
- 常用的归一化是log处理,之前离散程度很大的数据就被集中了;
- 常用的标准化是z-score:考虑到了不同样本对表达量的影响,消除到了表达的平均水平和偏离度的影响
它们的使用范围:
- 如果对表达量的范围有要求,用log归一化
- 如果表达量较为稳定,不存在极端最大最小值,使用归一化
- 如果表达量离散程度很大,存在异常值和较多噪音,用标准化可以避免异常值和极端值的影响
- 在分类、聚类、PCA算法中,使用z-score值的结果更好
- 数据不太符合正态分布时,可以使用归一化
- 机器学习的算法(SVM、KNN、神经网络等)要求归一化/标准化
绘制热图会经常用到z-score去除极端值
pheatmap(dat) # scale之前 n=t(scale(t(dat))) n[n>2]=2 # 限定上限 n[n< -2]= -2 # 限定下限 pheatmap(n,show_colnames =F,show_rownames = F) # scale之后接着:在单细胞分析中,也会同时用到Normalize和Scale(可以看: 单细胞Seurat包升级之2,700 PBMCs分析)
- 归一化 Normalize做的就是将数据进行一个转换,可以让同一基因在不同样本中具有可比性(例如RPKM、TPM等);另外降低离散程度。看使用的函数
LogNormalize背后的计算方法就是:log1p(value/colSums[cell-idx] *scale_factor),它同时考虑到了这两点- 标准化Scale就是基于之前归一化的结果(也就是log后的结果),再添z-score计算
最后,在 对细胞文库差异进行normalization 这一篇中也提到了:
- Normalization “normalizes” within the cell for the difference in sequenicng depth / mRNA thruput
- Scaling “normalizes” across the sample for differences in range of variation of expression of genes
- normalization一般是对文库处理,目的消除一些技术差异;scale一般对基因表达量处理(典型的z-score:表达量减均值再除以标准差),目的是后续分析不受极值影响
表达矩阵中的每个count值都表示成功的细胞捕获、成功的反转录、成功的测序。但即使是相同类型的细胞,它们的count depth(也就是每个细胞的全部表达量)也会有变化,变化的来源就在于上面说的那三步。因此在比较两个细胞时,任何差异都可能由于实验测序误差产生,而不是真的生物学差异。归一化就是解决这个问题,它把要比较的两个count值根据各自身处的环境求出一个相对丰度,也就是放在了一个水平上考虑,减少实验测序误差,突出更多的生物学差异。
最常用的归一化方法就是:count depth scaling,也称为counts per million(CPM),这个方法常用于bulk转录组,它会根据每个细胞的总表达量计算一个 size factor ,然后对其中各个基因表达量进行normalize。
这里再回顾下其他一些方法: 跟着豆豆一起回顾标准化方法 另外来自:https://cloud.tencent.com/developer/article/1484078
- RPM没有考虑转录本的长度的影响。适合于产生的read读数不受基因长度影响的测序方法,比如miRNA-seq测序,miRNA的长度一般在20-24个碱基之间
- RPKM/FPKM考虑了转录本的长度的影响。适用于基因长度波动较大的测序方法,如lncRNA-seq测序,lncRNA的长度在200-100000碱基不等
- TPM是先去除了基因长度的影响,而RPKM/FPKM是先去除测序深度的影响。TPM实际上改进了RPKM/FPKM方法在跨样品间定量的不准确性。
单细胞测序中使用的归一化方法由于细胞种类和基因错综复杂,有人就在bulk的基础上进行了改动。例如:Weinreb et al ( 2018) 先排除了表达量超过总体5%的基因,然后再计算size factor,主要是预防少量极高表达量基因的存在;Scran包有个pooling‐based size factor estimation方法,允许更高的细胞异质性存在;另外Scran包在批次矫正和差异分析环节也比其他归一化方法表现更好(Buttner et al, 2019)。
在单细胞RNA测序领域,目前有三种常用方法:其一是以10x Genomics为代表的微滴(droplet-based)测序;其二是以Namocell为代表的PCR板(plate-based)测序;其三是以BD Rhapsody为代表的微孔(micro-well-based)测序。就测序长度来说,Smart-seq/C1和Smart-seq2基于full length的测序方案,CEL-seq2, Drop-seq, MARS-seq, SCRBseq是基于UMI的测序方案。
不能指望某一种方法适用于所有类型的scRNA数据,(Cole et al, 2019)就发现不同的归一化方法对于不同类型数据集表现不同,使用scone工具可以帮助选择合适的方法。
一般在归一化后,数据都会变成log(x+1)的样子,但之后是否对基因进行z-score的标准化上,没有一个共识。Seurat的教程基本都使用了scale这一步,但Slingshot的作者就反对对基因进行scale (Street et al,
2018)。在本文中,作者倾向于避免对基因进行scale。
使用log转换的一个好处就是:让数据更加集中,减少数据的偏斜度,从而近似于许多下游分析工具对数据为正态分布的假设(尽管scRNA数据并不是真正的符合正态分布),比如在差异表达分析和批次矫正环节
小结
- 对于非全长scRNA数据(如10X),推荐使用 scran的归一化方法;
- 对于plate‐based 的数据,可以用 scone工具来进行评价,进而可以更好地处理plate之间的批次效应(Cole et al, 2019);
- 对于全长scRNA数据(如smart-seq)可以借用bulk的方法(如TPM),来矫正基因长度【这个问题在10X中不存在:in 10x single cell 3’ or 5’ gene expression assay, this gene-length bias does not exist. https://kb.10xgenomics.com/hc/en-us/articles/115003684783-How-to-calculate-TPM-RPKM-or-FPKM-instead-of-counts-】
- 归一化的数据应该是
log(x+1)这种形式的 - 是否进行scale没有共识,这里作者不推荐scale
4.4 数据矫正与整合
数据矫正的对象种技术和生物因素都有,例如:不同批次、捕获失败(dropout)、不同细胞周期。这些在之前的归一化中没有被矫正,但这些差异因素都可能会后面的分析产生影响,它们现在都是导致差异的”嫌疑人“之一。这里要做的就是把这些差异来源去掉(Regressing out 《=》【专门查的词典】 同义词partialling out :剔除)
4.4.1 首先是生物因素
最常见的生物矫正因素就是:转录组中的细胞周期信息。简单一点的方式就像Scanpy和Seurat对细胞周期评分进行简单线性回归;复杂点的方式就像scLVM和f‐scLVM。用来计算细胞周期分数的marker基因可以从文献中获得 (Macosko et al, 2015)。另外,这些方法还能用来去除其他已知的生物因素,例如线粒体基因表达量(可以作为细胞应激的标记)。
需要注意的是:
- 细胞周期因素并非一无是处,例如在一个增殖的细胞群中,所有细胞不是同步增殖的,那么就可以根据细胞周期评分来识别。所以需不需矫正还要根据研究目的判断
- 需要结合具体分析的生物问题来判断是否去除。生物体的多个生物过程往往存在依赖性,因此矫正其中一个过程,可能无意间掩盖了另一个过程
- 有人认为细胞大小的变化和细胞周期有关 (McDavid et al, 2016),因此在归一化过程中对细胞大小进行矫正,或者使用专用的工具如cgCorrect,也可以部分修正细胞周期的影响
4.4.2 然后是技术因素
最常见的技术矫正因素就是:样本测序深度、批次、噪音。
去除测序深度的影响,可以促进轨迹推断算法的表现,因为它需要在细胞之间找变化的路径,只要放在同一水平才能看到更准确的总体表达高低。
批次的来源可能是:细胞捕获的时期不同、文库制备使用的芯片不同、测序使用的lane不同。由此产生的效应存在于多个层面:一次实验中各个细胞群之间、同一实验室中进行的不同实验之间、或来自不同实验室的数据集之间。这里主要介绍第一种和最后一种情况:
- 第一种:一次实验中各个细胞群之间是最经典的情形,在bulk转录组也是常见的。使用线性方法进行矫正。例如ComBat工具就是利用线性矫正
- 最后一种:来自不同实验室的数据集之间的”数据整合“。使用非线性方法进行矫正。例如:CCA(Canonical Correlation Analysis)、MNN(Mutual Nearest Neighbours )、Scanorama、RISC、scGen、LIGER、BBKNN、Harmony。虽然这些非线性矫正方法也能用于第一种经典的批次情形,但可能会由于自由度增加而导致矫正过度。例如,在第一种经典模式下,Combat表现就比MNN好 (Buttner et al, 2019)
看一下Combat矫正前后的差别:其中颜色表示不同样本

去噪也是矫正的一种类型。单细胞数据的一个特点就是含有许多噪音来源,其中一个就是dropout。一些工具就用来推断dropout,用适当的表达量来替代0,例如:MAGIC、DCA、scVI、SAVER、scImpute。去噪可以提高基因间相关性的估计。这一步可以和归一化、批次矫正及其他下游分析整合起来,例如基于Python的 scVI工具。但任何方法都可能导致矫正过度或不足。
4.4.3 小结
- 判断要不要进行矫正生物因素:主要看后续分析是不是用于研究发育轨迹等特定生物过程
- 技术因素和生物因素需要放在一起矫正,而不是先矫正这个,后矫正那个
- plate-based数据的预处理一般需要利用非线性归一化方法
- 需要关注降噪前表达量为0,而降噪后才有表达的基因
4.5 挑选基因、降维、可视化
人类的scRNA数据中可能会包含25000个基因,但其中许多基因并非能提供有用信息,还有很多基因表达量直接为0。即使在QC阶段去掉这些表达量为0的基因,一个单细胞数据的基因空间依然会有超过15000个维度(一个基因表示一个维度),因此需要降低维度
4.5.1 首先挑选基因
就是挑那些真正”具有情报价值“的基因,也就是会数据变化起作用的基因。因此我们这里会挑选名为HVG的基因,也就是highly variable genes。根据数据集的复杂程度不同,HVGs一般会有1000-5000个(如下图就对不同数据集的HVGs做了个统计)
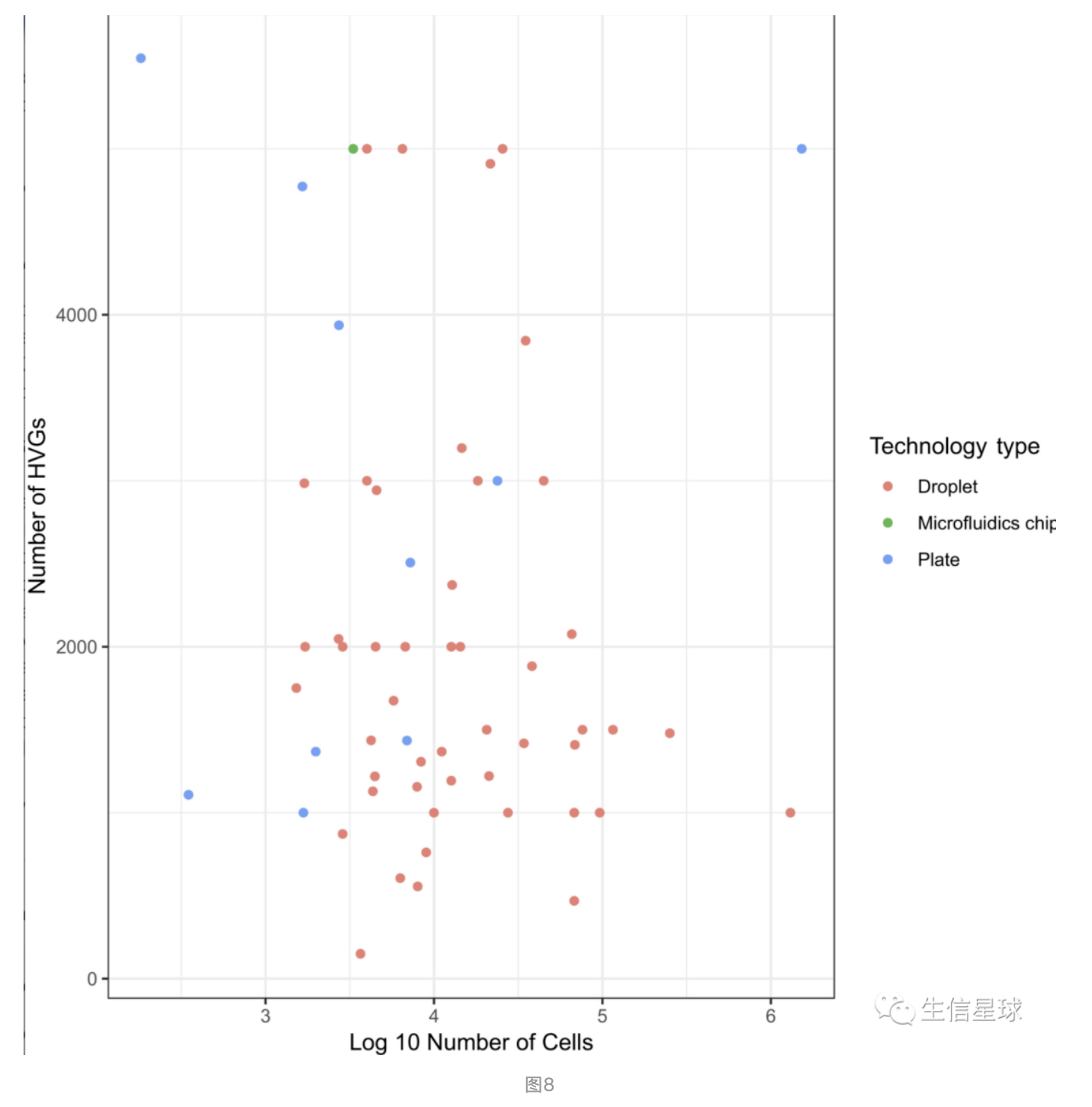
之前有研究表明,HVGs数量从200到2400,它们降维后的表现差不多(Klein et al ( 2015),作者建议先尽量多选一些HVGs。
比较流行的挑选HVGs的方法有Scanpy和Seurat,而且最好是在去除技术因素后挑选,避免因为批次、测序等因素导致错误挑选HVG。当然还有其他挑选的方法,看Yip et al ( 2018).
4.5.2 接着降维
挑出来HVGs后,就是降维了,力求在最少的维度中捕捉到最多的数据特征。
常用的降维方法:A-F分别是:PCA、t-SNE、diffusion maps、UMAP、ForceAtlas2(force‐directed graph)、Variance explained by the first 31 principal components (PCs)。关于单细胞数据的降维方法,详细可以看:Moon et al ( 2018)
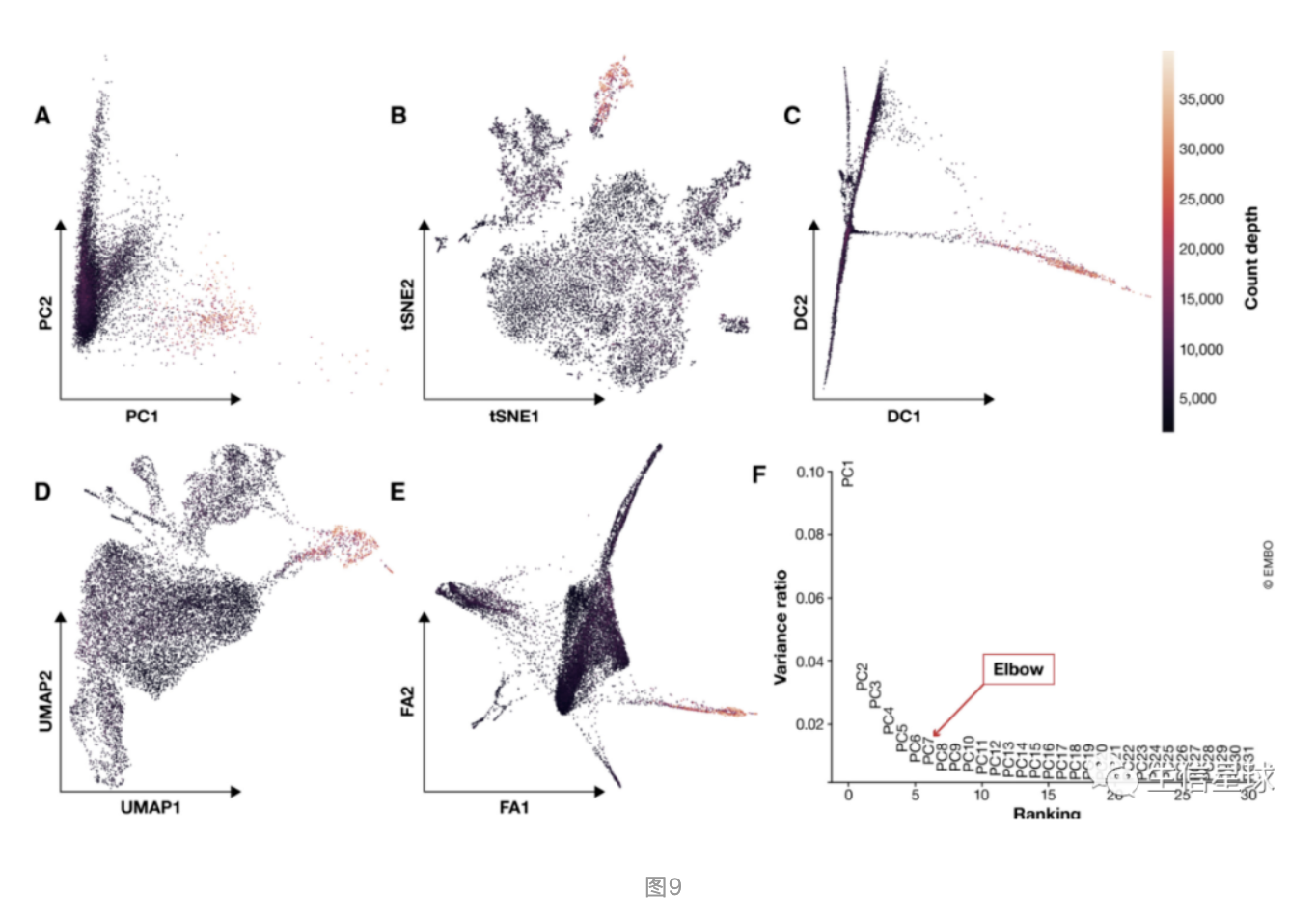
其中两个应用比较广的方法是:PCA(Pearson, 1901)和diffusion maps (Coifman et al, 2005) 【diffusion maps 于2015年在单细胞领域走红 Haghverdi et al ( 2015) 】
- 主成分分析PCA是一种线性方法,通过最大化每个其他维度中捕获的残差来生成缩减的维度。而且,PCA常作为非线性降维方法的预处理手段。PCA一般通过前N个主成分来表示整个数据集,其中N可以用F中的”肘elbow“部判断,或者用基于置换检验的jackstraw方法确定
- diffusion maps 是非线性的方法,它强调数据之前的转换。当研究连续型数据例如感兴趣的分化过程时会使用。它的每个成分(component)都能突出不同类型细胞间的异质性
4.5.3 最后可视化
可视化一般使用非线性降维的方法。最常用的就是2008年提出的t-SNE( t‐distributed stochastic neighbour embedding)。t-SNE的一个特性就是关注局部而忽视整体,因此带来的一个影响就是:可视化结果可能夸大了细胞群之间的差异,忽略了这些细胞群之间的潜在联系
另外,使用t-SNE的一大难点就是perplexity参数的设定,因为这个数不同,结果显著的cluster数也会不同 (Wattenberg et al,
2016)。
除了t-SNE,还有2018年推出的UMAP和SPRING可以用,在缺乏明确的生物学问题时,可以用UMAP作为不错的数据探索。
小结
- 根据数据集的复杂性,推荐选择1000-5000个HVGs
- 推荐UMAP进行数据探索;PCA获得一般性数据总结; diffusion maps作为PCA的替代,可用于轨迹推断
- PAGA方法与UMAP连用适用于特别复杂的数据集
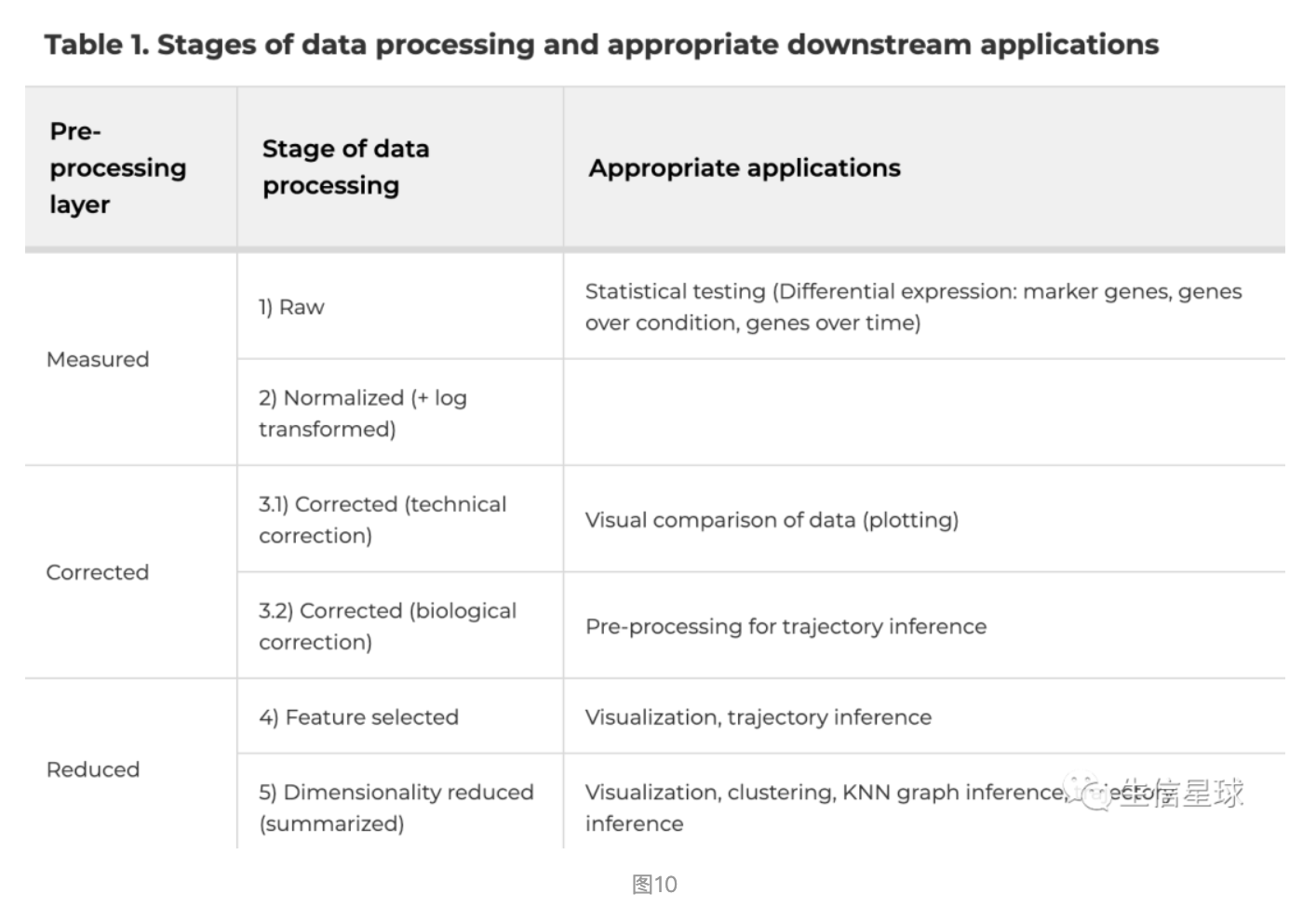
4.6 「总结」 预处理的各个阶段
作者贴心将预处理比作5种类型数据的处理:
原始数据(raw data)、归一化数据(normalized data)、矫正后的数据(corrected data)、挑选后的数据(feature‐selected data)、降维后的数据(dimensionality‐reduced data)
这5个阶段又分成3个层次:
- measured data:用于统计检验
- corrected data:用于数据比较可视化
- reduced data:用于下游分析
其中每个步骤适时调整,例如单一批次的数据集,就可以跳过矫正批次这一步
5 下游分析之细胞层面
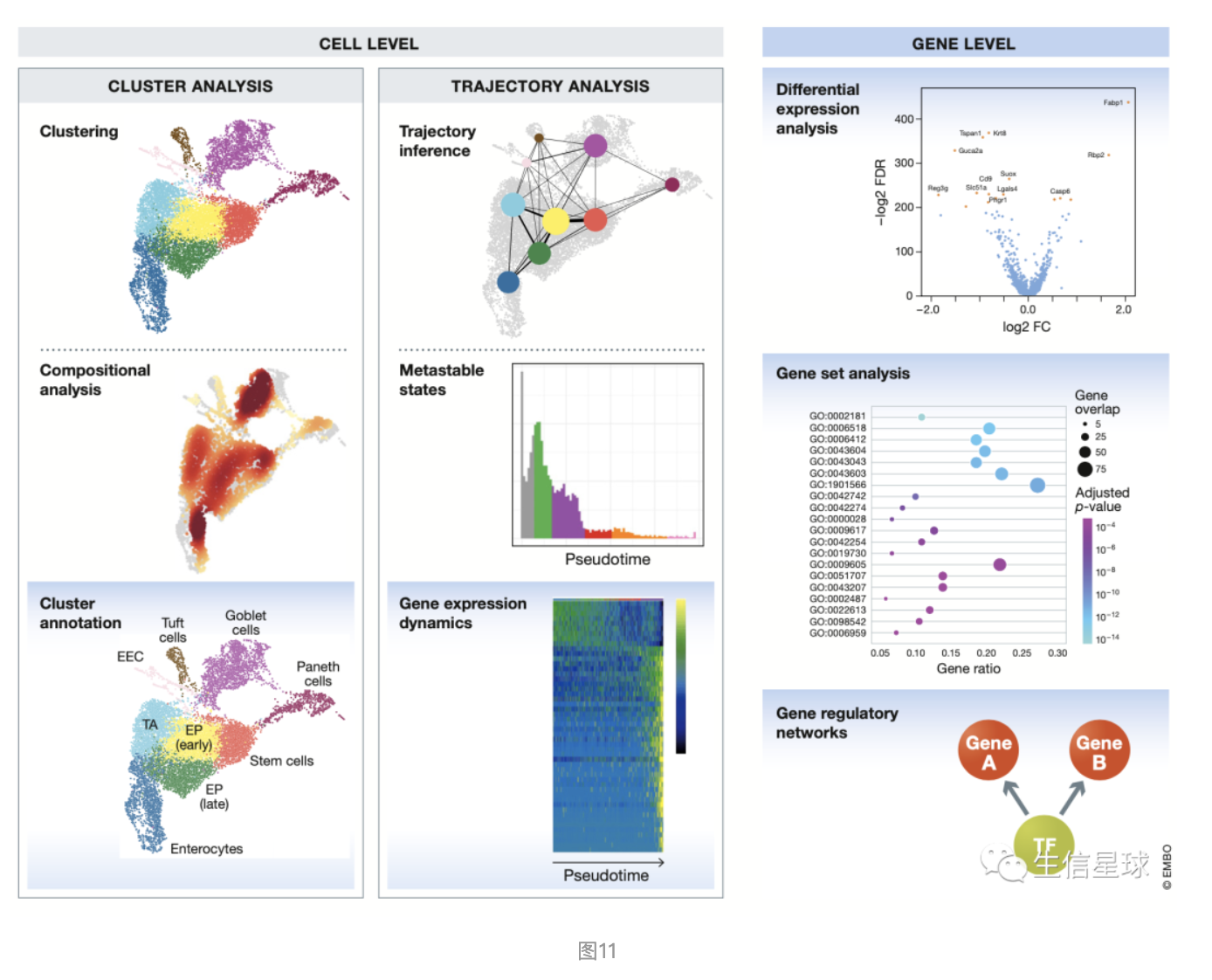
下游分析的目的是解释生物问题,例如根据表达量将细胞划分成不同的类型;相似细胞间表达量的微小变化也会体现连续的分化路径;基因表达量之间的相关性可能与基因共表达有关…
下游分析也是有细胞层面和基因层面:
- 细胞层面主要关注:分出多少群细胞、细胞的轨迹。细胞类型为了解释异质性的问题;轨迹作为一个动态发育过程中的一个”快照“,可以帮助理解某个动态发育过程
- 基因层面就是:差异分析、富集分析、互作网络
下面👇先看看看细胞层面的分析之分群和轨迹
5.1 细胞分群
5.1.1 先是:分群方法
这里主要都是算法相关,简单了解即可
将细胞分群基本就是任何单细胞分析的必经之路。群的划分就是根据细胞中基因表达谱的相似性,表达谱的相似性是由于欧几里得距离量度决定的,而距离量度又是利用的降维的数据。一般有两种方法计算:clustering algorithms、community detection methods
clustering algorithms是直接基于距离的经典无监督机器学习方法。最常见的是k-means。k-means使用的空间距离量度也有不同(默认是欧氏距离),比如cosine similarity、 correlation‐based distance metrics、the SIMLR method。最近的研究表示correlation‐based distances优于其他距离 (Kim et al, 2018)。
community detection methods是graph‐partitioning algorithms,利用了K近邻法 K‐Nearest Neighbour approach (KNN graph)作图。细胞就是图上的一个节点,每个细胞都和K个最相似的细胞连接,这个相似性也是根据降维后空间中的欧氏距离计算的。根据数据集的大小,K一般设为5-100
community detection methods一般比clustering algorithms速度快,因为只有相邻的细胞对被认为属于相同的群,大大减少了可能的细胞群的搜索范围
5.1.2 然后是:分群后的注释
这个过程主要是基因层面的操作,为每个cluster找marker gene(也就是能代表这个cluster的基因,而这个基因又和已知的细胞类型有关)。任何的分群算法和参数设置都会将一整团细胞分成多个群,但这些群是否真的有意义,就要靠这一步来和生物背景结合起来。
我们希望看到的是存在很多类型的细胞,来说明细胞异质性的问题,但这里关于细胞类型这个定义还是存在争议。首先,细胞类型的划分怎样算是清楚,对于一些人来说,”T cells“这个名称可以叫一个细胞类型,但还有人认为,必须继续深入,像”CD4+ T cells“、”CD8+ T cells“才叫细胞类型;另外,即使是同一种细胞类型的细胞也会有不同的发育状态,因此它们也会显示不同的分群结果。但不管如何,它们都是当时细胞的一种身份(identity)
这个很好理解,就像人一样,人生阶段不同身份也不同,但不能简单说它的类型发生了变化。
因此,我们将分群的结果称为不同身份的细胞(cell identities)会比不同类型的细胞(cell types)要好一些【即每个亚群可能并不是真的不同类型细胞,只是显示了此时此刻的细胞身份】
对于不同细胞身份的注释,近年来也随之细胞图谱的研究而加速,例如小鼠脑细胞图谱 (Zeisel et al, 2018) 、人类细胞图谱 (Regev et al, 2017)的发现,产生了许多参考数据库。在缺乏相关背景的情况下,我们可以借用数据库中已发现的细胞marker 基因套入我们的细胞,帮助判断细胞身份。需要注意:通常使用的细胞表面marker基因在细胞身份鉴定方面存在局限性(Tabula Muris Consortium et al, 2018)
看这个注释结果: 图A是利用Louvain方法分群+UMAP可视化; 图B是细胞身份的鉴定:stem cells (Slc12a2), enterocytes (Arg2), goblet cells (Tff3) and Paneth cells (Defa24)。但要注意marker基因可能也会在其他身份的细胞中表达,例如很多marker都在右上角(goblet and Paneth 细胞)中有表达,但最后还是根据表达量来指定特定的细胞身份(比如Slc12a2基因虽然在很多细胞都表达,但就是在中部偏右这一坨细胞中表达量相对高,所以把它当做stem cell) 图C是近端(上图)和远端(下图)肠上皮区域的细胞身份组成图(颜色越深细胞密度越大)
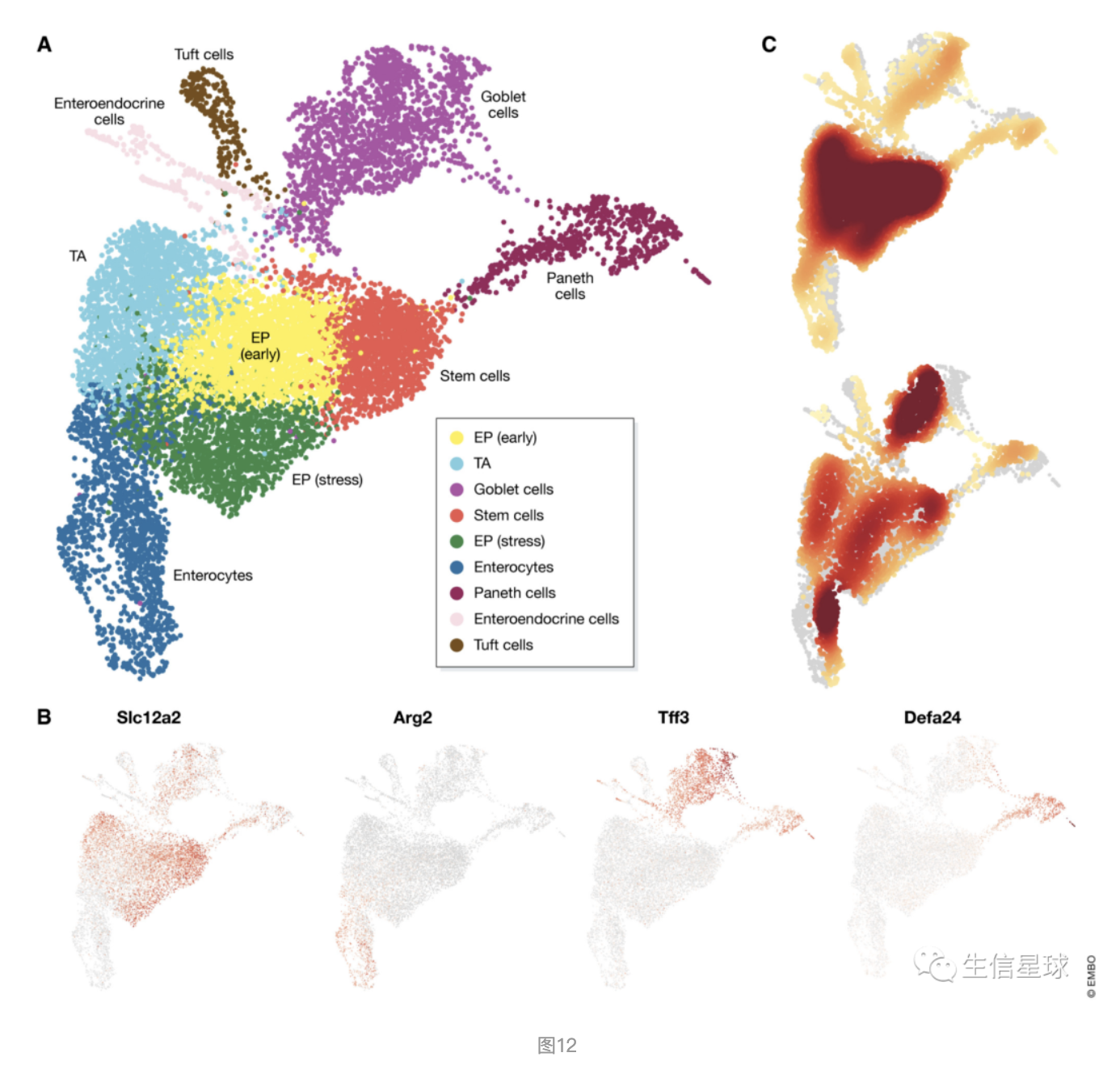
上面提到根据marker基因进行细胞分群注释。那么marker基因怎么获得?
利用差异分析,分成两组:某个cluster中的细胞、数据集中其余全部的细胞。然后重点关注这个cluster中上调的基因,因为marker基因一般具有更强的表达作用。差异分析也会使用简单的统计检验,例如Wilcoxon rank‐sum test、t-test,将基因的差异大小排个序,选出排名靠前的基因来作为marker基因
有了marker基因,再进行注释
将数据集中选出的marker基因和参考数据集进行比对,统计方法可以是:enrichment tests、the Jaccard index、other overlap statistics
参考数据集可以是网页工具: www.mousebrain.org、 http://dropviz.org/,可以将选出的marker基因在参考数据集中进行可视化,帮助判断这个marker基因是什么细胞身份
注释并非一蹴而就,这个很麻烦…
细胞分群、分群注释、重分群、重注释…这个循环很耗费时间。自动化注释方法加快了这个过程,例如scmap (Kiselev et al, 2018b) 、Garnett (preprint: Pliner et al, 2019) ,但这样的方法有利有弊。自动化提高了速度,但相比手动注释也降低了灵活性。毕竟自动化工具使用的参考数据集中可能并不包含我们数据中的这样细胞。因此,有自动化工具也不能完全抛弃手动挑选,尤其针对大型数据集中多种多样的细胞。自动化的过程可以先帮我们粗略地给细胞加个标记,如果有需要,我们可以继续手动对这种细胞继续划分子细胞。对于小型数据集或者缺乏参考基因集的,手动注释就足够了。
5.1.3 注意
- 同一细胞身份的marker基因在不同数据集之间可能由于数据集细胞类型和状态组成而不同【选出的marker基因并不是说以后遇到它,就一定等同于这种类型的细胞。只是说在某种细胞的某个状态下,这个marker基因更符合】
- 如果存在参考数据集(例如 www.mousebrain.org、 http://dropviz.org/),建议辅助自动化工具进行注释,减少手动查基因的时间
5.1.4 细胞分群衍生——细胞组成分析(Compositional analysis)
就像上面的图12中的C图,显示的是近端(上图)和远端(下图)肠上皮区域的细胞身份组成图(颜色越深细胞密度越大)。研究细胞组成的变化也是一个新方向,例如沙门氏菌感染已被证明会增加小鼠肠上皮细胞的比例 (Haber et al, 2017)。
这个分析既需要足够多的细胞数量来推断各个cluser的占比,又需要足够的样本数量来证明是单纯一个样本得cluster数量这样变还是总体都会这样变。相关的分析工具还没有太多,未来的开发可能会借鉴单细胞质谱流式(mass cytometry)或者是宏基因组分析【单细胞与宏基因组的结合…】
5.2 轨迹分析
5.2.1 轨迹推断Trajectory inference
轨迹推断就是为了找到不同细胞身份、分化或者生物过程中渐进式非同步的变化,构建出的一个动态模型。它认为单细胞数据实际上就是一个连续过程中的快照(snapshot),这个过程可以通过在细胞空间中寻找最小化相邻细胞间转录变化的路径来重建
例如: 图A就是利用Slingshot推断近端(proximal)和远端(distal)肠上皮细胞的分化轨迹; 图B就是在PCA空间中进行的Slingshot推断。图中细胞的路径就叫做”拟时序“(pseudotime)
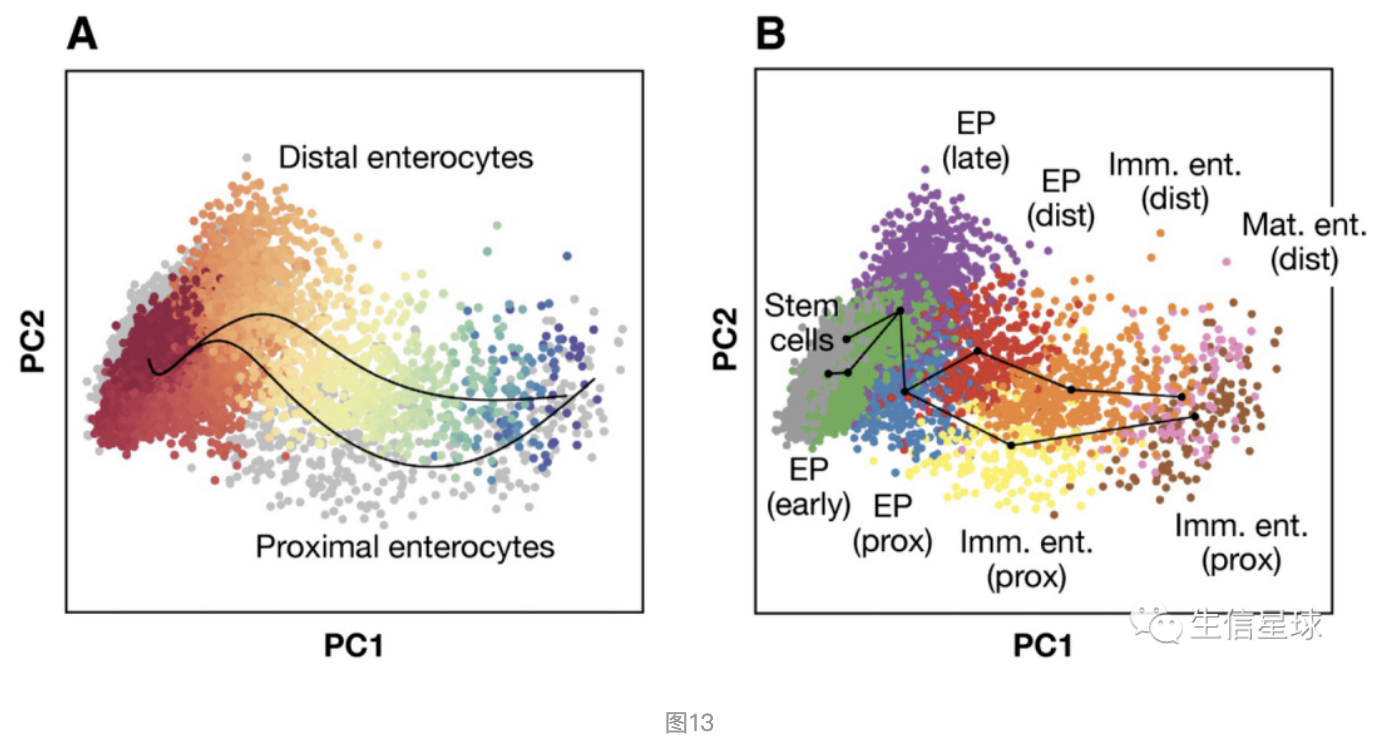
2014年Monocle和Wanderlust先推出了轨迹推断,之后诞生的分析方法更加丰富,它们在建模路径的复杂性上有所不同,从简单的linear or bifurcating(分叉) trajectories,到复杂的graphs, trees, or multifurcating(多叉) trajectories。Saelens et al, 2018)进行过轨迹推断方法的比较,结论是没有一种方法对所有类型的轨迹推断有效,应该根据预期轨迹的复杂度来选择。不过,Slingshot在简单轨迹推断中优于其他方法(Street et al, 2018) 。如果期望得到更复杂的轨迹,PAGA值得推荐。轨迹推断是一个不确定的过程,可以用多种方法来进行佐证。
细胞内通常会同时发生多个生物学过程,因此在进行发育轨迹推断时,可以将其他生物因素去掉,例如T细胞在逐渐成熟的过程中就可能会经历细胞周期转变(Buettner et al, 2015)。
另外轨迹推断最好是在细胞分群之后进行,因为一个cluster的形成可能意味着这一坨细胞处于比较稳定的状态了。
此外,RNA速率(RNA velocities)可以添加发育轨迹的方向,例如: scVelo
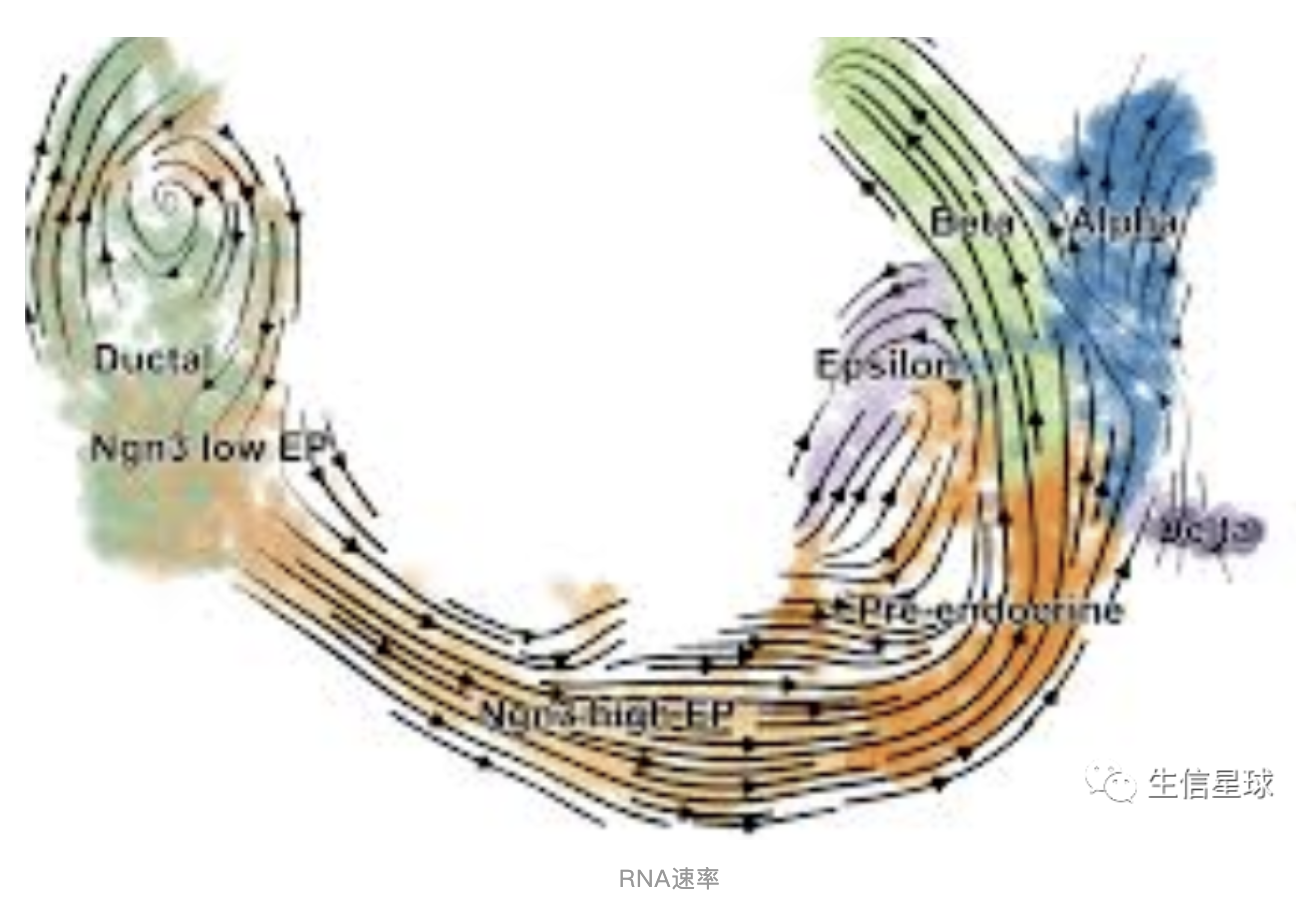
当然,推断的轨迹不一定就代表一个生物学过程,因为毕竟是根据”快照“数据中的转录状态推测的。后续可以借鉴:perturbation experiments、inferred regulatory gene dynamics、support from RNA velocity
5.2.2 基因表达量的动态变化
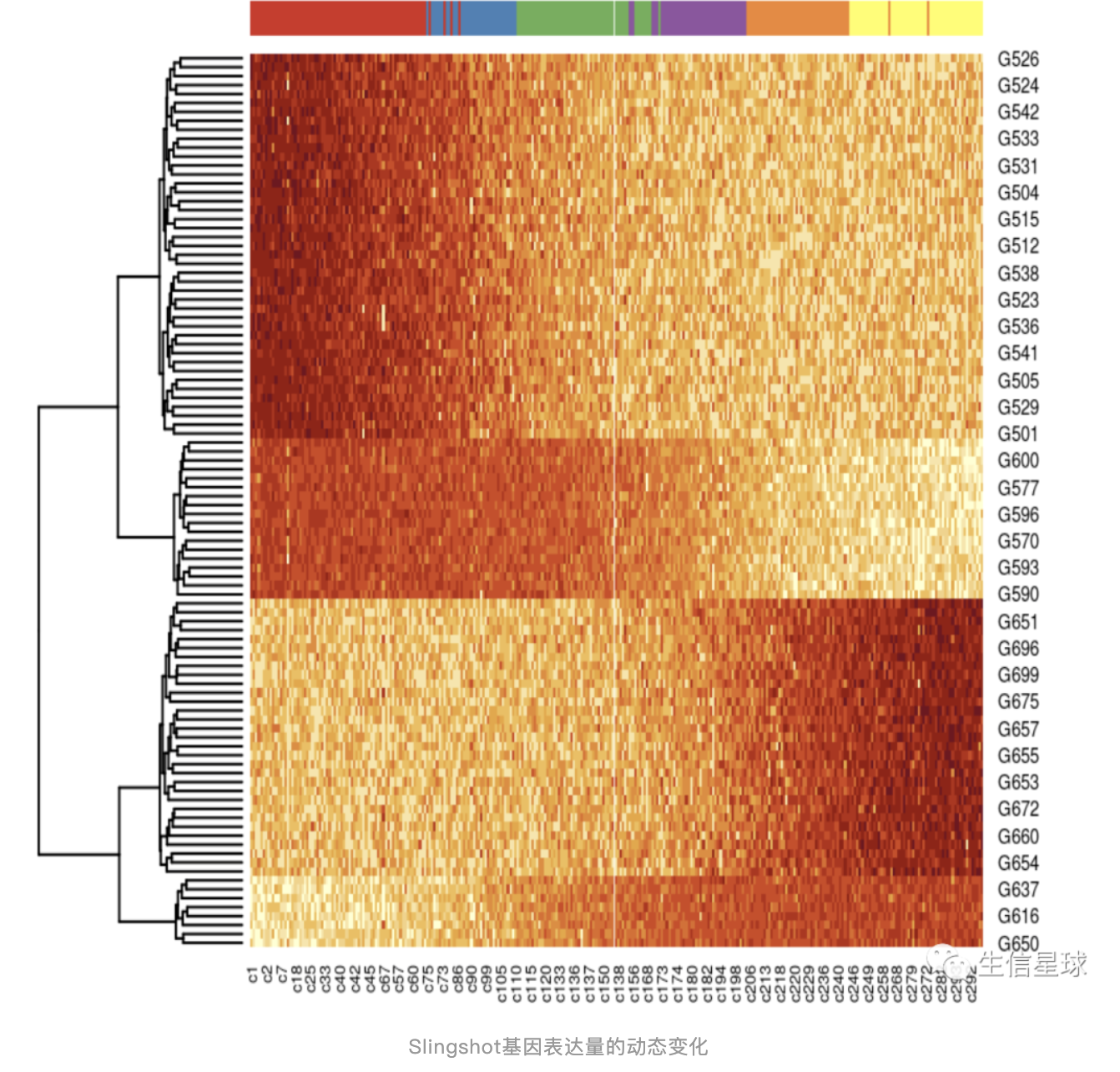
在拟时序(pseudotime)中变化的基因描述了轨迹,这组与轨迹相关的基因有望包含调控建模过程的基因,可以用来识别潜在的生物过程。
目前很少有专门分析基因表达动态变化的工具。BEAM将Monocle的轨迹推断整合进来,允许检测在轨迹分支过程中相关基因的动态变化。另外还有LineagePulse (https://github.com/YosefLab/LineagePulse)考虑了dropout技术噪音但还在开发中。
下面这样的图在Slingshot的帮助文档就有提及:https://bioconductor.org/packages/release/bioc/vignettes/slingshot/inst/doc/vignette.html 【4.1:Identifying temporally expressed genes】
require(gam)
t <- sim$slingPseudotime_1
# for time, only look at the 100 most variable genes
Y <- log1p(assays(sim)$norm)
var100 <- names(sort(apply(Y,1,var),decreasing = TRUE))[1:100]
Y <- Y[var100,]
# fit a GAM with a loess term for pseudotime
gam.pval <- apply(Y,1,function(z){
d <- data.frame(z=z, t=t)
suppressWarnings({
tmp <- suppressWarnings(gam(z ~ lo(t), data=d))
})
p <- summary(tmp)[3][[1]][2,3]
p
})
topgenes <- names(sort(gam.pval, decreasing = FALSE))[1:100]
heatdata <- assays(sim)$norm[topgenes, order(t, na.last = NA)]
heatclus <- sim$GMM[order(t, na.last = NA)]
heatmap(log1p(heatdata), Colv = NA,
ColSideColors = brewer.pal(9,"Set1")[heatclus])
5.2.3 细胞亚稳态分析 Metastable states
亚稳态常见于物理化学。在物理学中,亚稳性(Metastable)是动力系统的一种稳定状态,而不是系统能量最低的状态 Metastable:stable provided it is subjected to no more than small disturbances. 另外这个状态可以用这个图帮助理解:大概就是「一个相对稳定但又会变化的一个状态」
拟时序分析会展示出不同阶段细胞数量的多少。假设细胞以无偏的方式采样,其中轨迹中的稠密区域就表示转录时首选的方案。当把轨迹理解为一条时间线时(例如在发育这个时间线),这些密集的区域可能代表细胞的亚稳态,可以结合拟时间坐标来绘制直方图,找到这些亚稳态【因此看到B图中很多种状态,但C中直方图认为这几个密集的区域才属于亚稳态】
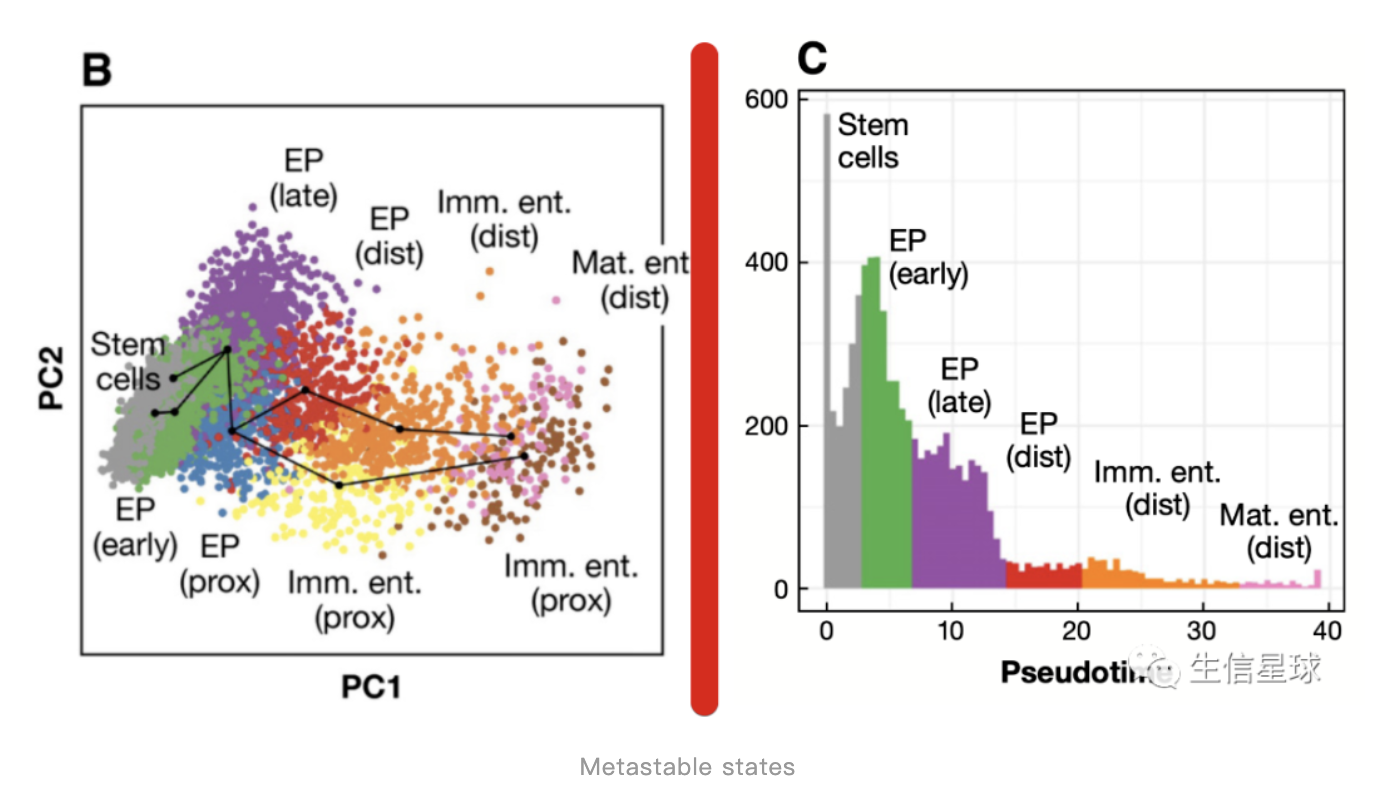
5.2.4 整合分群与轨迹分析
分群是由整体到部分,是静态的;而轨迹又是由部分推断整体,是动态的。二者结合起来又产生了一种新的分析模式
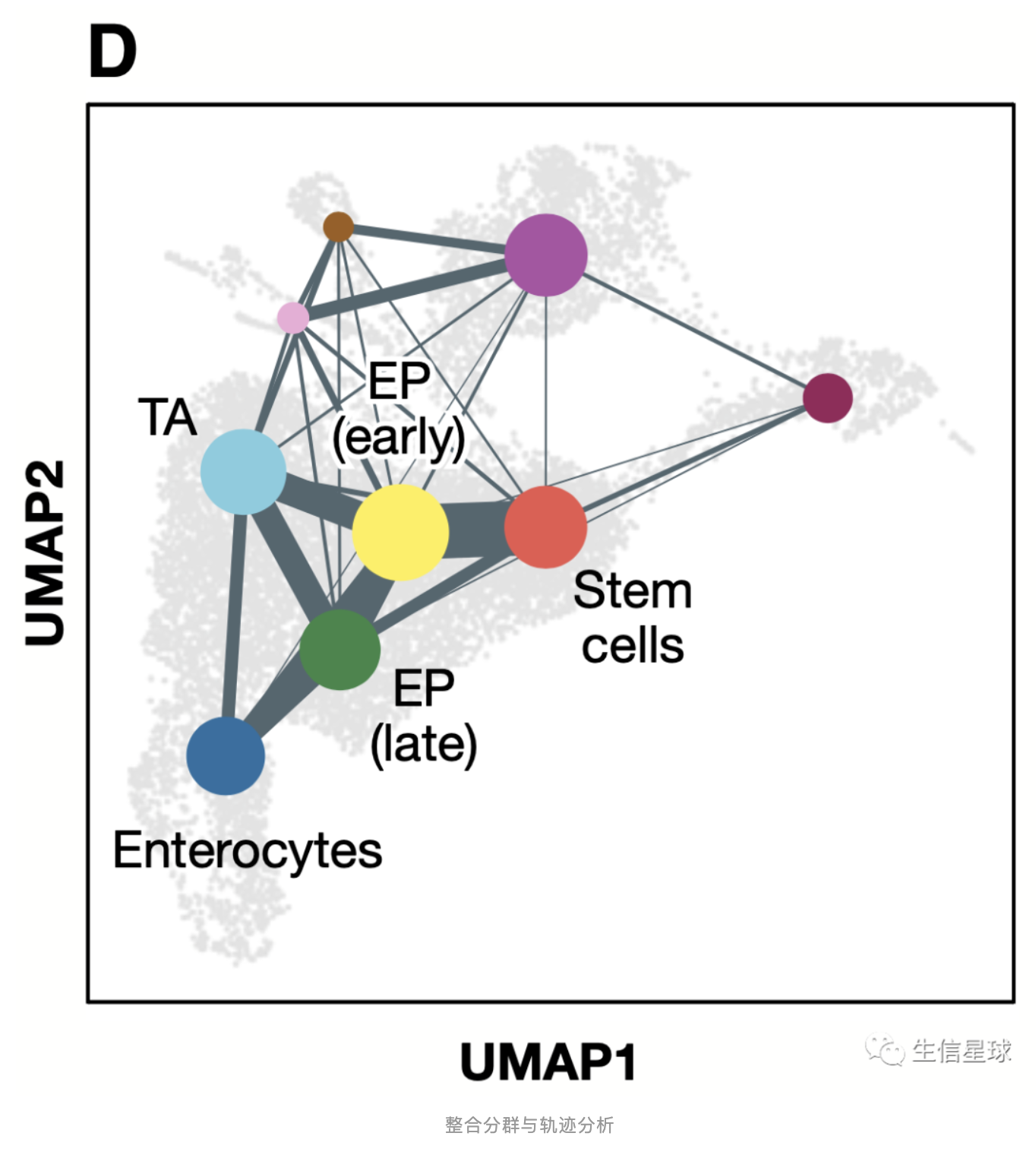
将分群的结果当成节点(node),将轨迹当成节点之间的桥梁(edge),所以将动静数据结合在了一起。利用partition‐based graph abstraction(PAGA)这个工具就能得到类似下面这个图。
It was the only reviewed method able to cope with disconnected topologies and complex graphs containing cycles.
6 下游分析之基因层面
之前都是对细胞进行分析,但细胞中的基因分析会提供更多的信息。例如差异表达分析、基因集分析和基因调控网络推断,不是表面上研究细胞异质性,而是基于异质性探索基因表达相关的原因
6.1 差异表达分析
基因层面的数据探索,一个经常遇到的问题就是:两个组之间有没有表达量的差异?
这个方法也是常规bulk转录组中经常做的。不过单细胞相比于bulk转录组的一个优势就是:可以深入一个层次,原来bulk只是看一块组织的平均表达量,但现在经过分群后,能得到一块组织中各种各样的亚群,再结合差异分析,对理解异质性问题更有帮助。
虽然都是朝着一个方向前进,但单细胞和bulk转录组的差异分析方法还是不同的。
- bulk转录组存在样本数量的限制,因此算法需要对少量样本进行准确估计,而单细胞则不同,一个细胞作为一个样本,成百上千不在话下;
- 单细胞数据又有自己的特点:特异的人为技术噪音(dropout、high cell‐to‐cell variability ),因此单细胞分析方法需要额外考虑这些因素
但最近(Soneson & Robinson, 2018)研究表明,基于大批量的差异分析,bulk分析方法的性能与最好的单细胞分析方法相当。当bulk方法进行改进,加入基因权重分析后,表现要好于单细胞原有工具。例如:bulk差异分析工具DESeq2/EdgeR + ZINB‐wave工具估算的权重。
不过,bulk差异分析工具的性能虽然好,但是计算的效率很难提升。毕竟单细胞数据样本数量越来越多,程序跑的时间长短也成了衡量工具优劣的重要因素。单细胞工具MAST脱颖而出。在单个数据集的小范围比较中,完胜bulk和其他单细胞方法(Vieth et al, 2017)。而且MAST比bulk方法快了10到100倍 (Van den Berge et al, 2018) 。
小结
- 差异分析使用MAST或limma
- 差异分析不能使用矫正后的数据(denoised, batch corrected, etc.),而是应该在计算过程中去指定需要矫正的技术因素
- 我们给差异分析算法提供的矫正的因素(称之为协变量covariates)不能太混乱,因为工具不会去智能识别,必须要清楚需要矫正什么
6.2 基因集分析
基因层面的分析,往往会产生大量的基因,但很难去解释。
例如差异分析我们往往能得到上千基因,为了比较方便解读,一般会把有共同特性的基因归为一组,然后检查我们归类的可靠性 【grouping the genes into sets based on shared characteristics and testing whether these characteristics are overrepresented in the candidate gene list.】
我们一般关注基因在生物过程(biological processes, BP)中的富集,可以使用MSigDB、GO、KEGG pathway、Reactome数据库
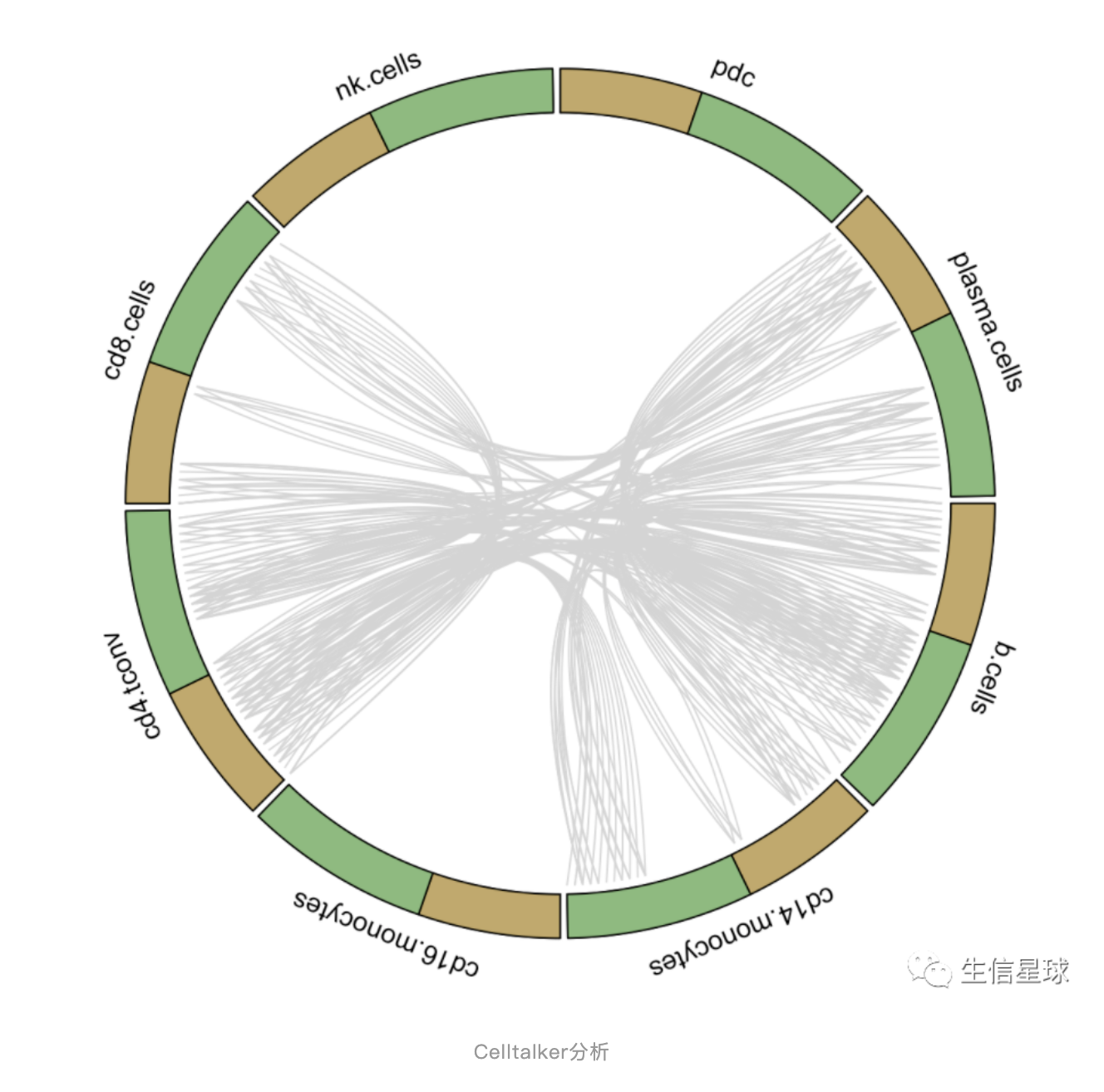
另外,单细胞中的一个新进展就是利用成对基因标签进行配体受体分析( ligand–receptor analysis)
来自:https://www.bio-equip.com/showarticle.asp?id=453107210 肿瘤内细胞-细胞相互作用的研究将通过对配体和受体的表达分析来探究细胞间的相互交流。运用配体-受体复合物的数据库,通过scRNA-seq数据与肿瘤细胞亚群的定义相结合,来推断潜在的细胞-细胞相互作用,可以理解为其中一个群体产生配体,向另一个表达相应受体的群体发信号
配体-受体成对标签可以从:CellPhoneDB数据库获得,然后用来解释cluster之间高表达基因的联系
例如,利用 celltalker 就可以做
6.3 基因调控网络 gene regulatory network (GRN)
基因并非独立发挥作用的。相反,基因的表达水平取决于与其他基因和小分子之间的相互调控
方法例如: SCONE、 PIDC、 SCENIC (Single-Cell rEgulatory Network Inference and Clustering),但发展还不是很完善,推断的调控关系不是很稳定【谨慎使用】
7 分析平台
现在开发了很多平台,整合了一套分析流程,有基于R的(McCarthy et al, 2017; Butler et al, 2018) ,python的 (Wolf et al, 2018),本地的(Patel, 2018; preprint: Scholz et al, 2018) ,网页版带可视化的(Gardeux et al, 2017; Zhu et al, 2017)
Seurat是使用最广泛的,Scater在QC和预处理中表现优异;除此以外,基于Python的scanpy也逐渐发展起来,它对于大量细胞的标准化方面表现不错
如果不使用命令行,可视化界面也有,只不过用户只能跑人家已经写好的脚本,操作灵活性不足。这样的平台更多的用处是在可视化探索上,例如Granatum、ASAP。未来 Human Cell Atlas(HCA)会在数据可视化探索上迅速发展: https://www.humancellatlas.org/data-sharing
8 结语
8.1 作者的结语
作者把流程测试和说明都放在了:https://github.com/theislab/single-cell-tutorial
感兴趣的可以跟着走一遍,比较一下不同的工具。作者希望这一篇能代表单细胞领域目前发展的一个最新动向。他也提到,新方法层出不穷,本文介绍的大量的方法是经过实践比较、验证过的。**目前可用的方法不管是运行效率还是易用性可能都不如最新开发的方法,但要注意:新方法在未被大量验证之前都需小心使用。**而且新方法一般都是针对单个层面(比如降维、分群、轨迹推断等),大体的分析流程基本固定了。
未来整合深度学习和单细胞多组学是两个重要的发展方向,流程化运行更是趋势。
随着文库制备和测序技术的进步,未来的单细胞平台必将可以处理多种类型数据:DNA甲基化、蛋白丰度等等。
8.2 刘小泽的结语
截止到2020年3月8日下午15.35,打卡看完!
三天的时间,基本每天都会花半天时间在阅读这篇综述上。从第一眼看到它的文章逻辑,就感觉:嗯是它,没错了!连午觉都不想睡了。
一开始想强迫自己看下去,没想到,越看越精彩。尤其是将整个流程和自己的知识结合起来,就看得比较顺畅。为了更加易读,我在其中加了很多注释,包括之前自己写的一些推文和网上一些好的资源,可以帮助梳理知识点。
最后,希望看完本文对你有帮助🤓!