scRNA-10X免疫治疗学习笔记-4-细胞亚群的生物学命名
刘小泽写于19.10.15 笔记目的:根据生信技能树的单细胞转录组课程探索10X Genomics技术相关的分析 课程链接在:http://jm.grazy.cn/index/mulitcourse/detail.html?cid=55 第二单元第8讲:细胞亚群的生物学命名
上一次我们好不容易得到了这个1.9G的RData,也就是作者自己做出来的Seurat对象,那么我们要怎么利用它去进一步探索呢?
加载上次运行的结果
首先还是三步走
rm(list = ls())
options(warn=-1)
suppressMessages(library(Seurat))
然后加载进来之前保存的5.1G PBMC对象
start_time <- Sys.time()
load('./patient1.PBMC.output.Rdata')
end_time <- Sys.time()
end_time - start_time
# Time difference of 12.6741 secs
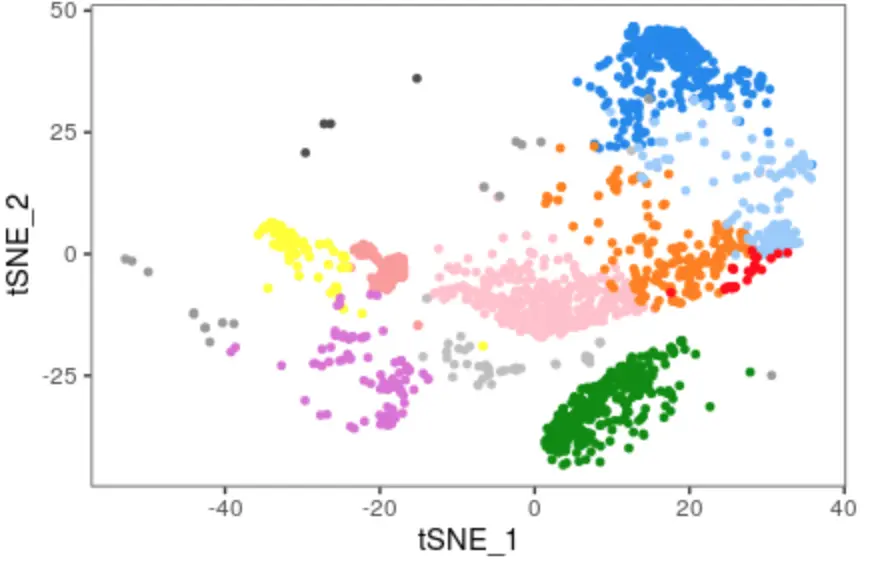
重温上次的聚类结果
# 使用Seurat 2.3.4版本
colP<-c('green4',
'pink',
'#FF7F00',
'orchid',
'#99c9fb',
'dodgerblue2',
'grey30',
'yellow',
'grey60',
'grey',
'red',
'#FB9A99',
'black'
)
TSNEPlot(PBMC,
colors.use = colP,
do.label = T)

开始新的探索
看看作者整合的数据对批次的处理
也就是把四个时间点映射到上面的tsne坐标中,并且理论上应该是:每群细胞都覆盖到四个时间点
TSNEPlot(PBMC,group.by = "TimePoints")
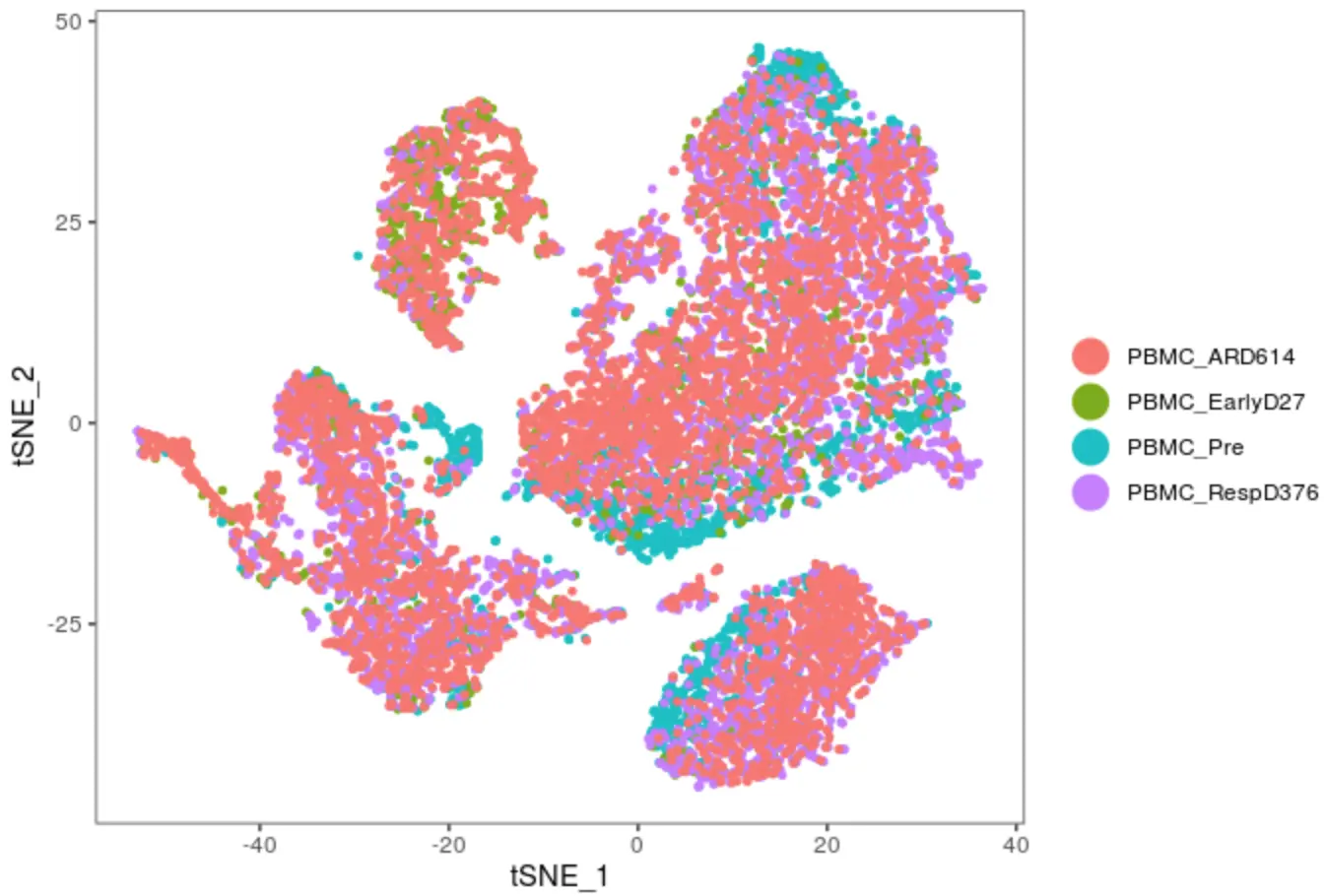
再用table对比一下
> table(PBMC@meta.data$TimePoints,PBMC@ident)
0 1 2 3 4 5 6 7 8 9 10 11 12
PBMC_ARD614 665 726 572 559 420 302 457 313 283 123 17 11 68
PBMC_EarlyD27 43 173 245 85 120 110 543 59 91 29 7 3 84
PBMC_Pre 369 527 197 93 146 393 4 76 17 48 25 187 0
PBMC_RespD376 800 433 555 677 636 516 119 324 204 200 170 11 39
可视化一些marker基因
这些marker基因也是来源于文章的Supp Fig.7,基于他们对免疫知识的了解
allGenes = row.names(PBMC@raw.data)
markerGenes <- c(
"CD3D",
"CD3E",
"TRAC",
"IL7R",
"GZMA",
"FCGR3A",
"CD14",
"MS4A1",
"FCER1A"
)
# 判断这些marker是不是存在表达矩阵中
> markerGenes %in% allGenes
[1] TRUE TRUE TRUE TRUE TRUE TRUE TRUE TRUE TRUE
# 选择性保存pdf格式
# pdf('patient1_pBMC_marker_FeaturePlot.pdf', width=10, height=15)
FeaturePlot(object = PBMC,
features.plot =markerGenes,
cols.use = c("grey", "blue"),
reduction.use = "tsne")
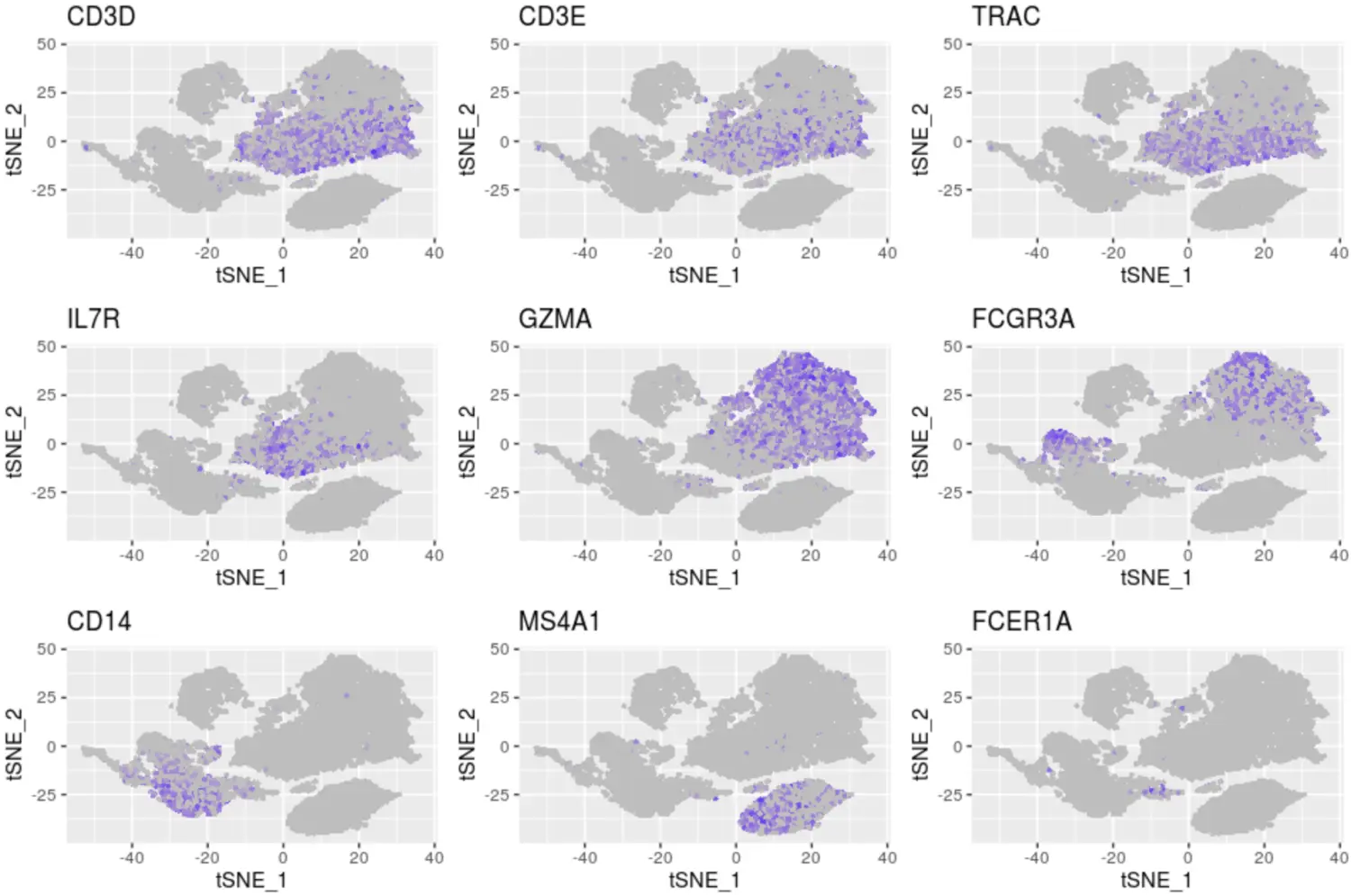
利用上面marker基因在不同细胞群的特殊表达,其实就能得到每个群的细胞命名,不过这些marker基因的获得以及和细胞名称的对应,是需要一段时间的研究才能得到的。我们这里只是展示如何操作
赋予每个cluster细胞类型
根据我们得到的cluster和原文的命名
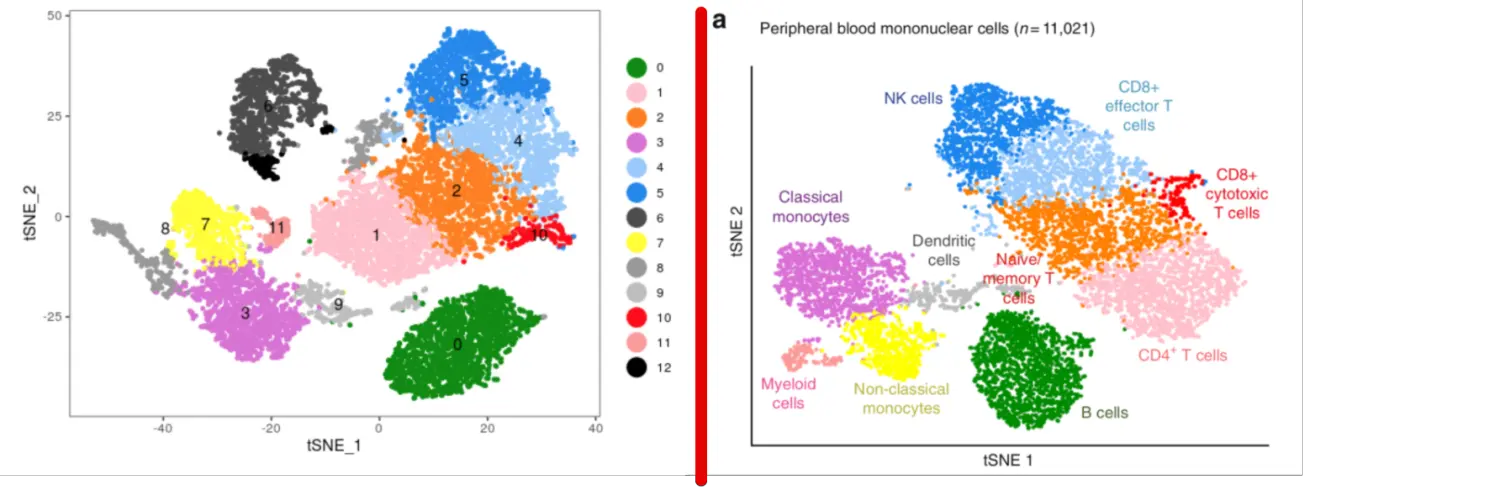
就可以对应得到一个细胞名称列表:
cat >celltype-patient1-PBMC.txt
0 "B cells"
1 "CD4+ T cells"
2 "Naive memory T cells"
3 "Classical monicytes"
4 "CD8+ effector T cells"
5 "NK cells"
6 "rm1"
7 "Non-classical monocytes"
8 "Dendritic cells"
9 "rm2"
10 "CD8+ cytotoxic T cells"
11 "Myeloid cells"
12 "rm3"

假如我们是先有了这个列表,核心就是要将分群的clsuter数字与细胞名称和颜色对应起来
利用Seurat V3
版本3的优势是可以直接提取出seurat对象的cluster数字,并且提供了RenameIdents函数,直接进行转换
a=read.table('celltype-patient1-PBMC.txt')
new.cluster.ids <- as.character(a[,2])
names(new.cluster.ids) <- levels(PBMC_V3)
PBMC_V3 <- RenameIdents(PBMC_V3, new.cluster.ids)
DimPlot(PBMC_V3, reduction = "tsne", label = TRUE, pt.size = 0.5, cols = colP) + NoLegend()

利用Seurat V2
版本2就需要自己去对应:首先要了解它的分群信息存储在PBMC@ident中,然后要将全部12874个细胞的分群编号与celltype-patient1-PBMC.txt表中的第一列编号对应(只有这样,才能和表中的第二列对应上)
用到对应关系时,首先思考
match能不能做到;如果要做,需要准备什么;对应关系搞清楚
# 先与表中第一列对应
match(as.numeric(as.character(PBMC@ident)),a[,1])
# 第一点需要注意的是:为什么先用as.character后用as.numeric,而不是直接用as.numeric?
# 原因就是PBMC@ident存储的是因子型变量,直接取只会得到它们的位置信息,而不是真实的分群信息
> head(as.numeric(PBMC@ident))
[1] 2 1 2 2 2 6
> head(as.character(PBMC@ident))
[1] "1" "0" "1" "1" "1" "5"
> head(as.numeric(as.character(PBMC@ident)))
[1] 1 0 1 1 1 5
# 第二点需要注意的是:match函数的规则是,A要在B中找到对应位置,那么就是 match(A,B)
接下来就可以得到对应的第二列,也就是细胞名称
labels=a[match(as.numeric(as.character(PBMC@ident)),a[,1]),2]
# 检查一下,主要看数量的对应
> table(labels)
labels
B cells CD4+ T cells CD8+ cytotoxic T cells
1877 1859 219
CD8+ effector T cells Classical monicytes Dendritic cells
1322 1414 595
Myeloid cells NK cells Naive memory T cells
212 1321 1569
Non-classical monocytes rm1 rm2
772 1123 400
rm3
191
> table(PBMC@ident)
0 1 2 3 4 5 6 7 8 9 10 11 12
1877 1859 1569 1414 1322 1321 1123 772 595 400 219 212 191
添加到metadata中,方便后面使用
PBMC@meta.data$labels=labels
TSNEPlot(PBMC, group.by = 'labels',
colors.use = colP,
do.label = T)
但很明显,颜色标记和原文不同。因为之前只是对应了分群的编号和细胞名称,此外还需要修改颜色的顺序
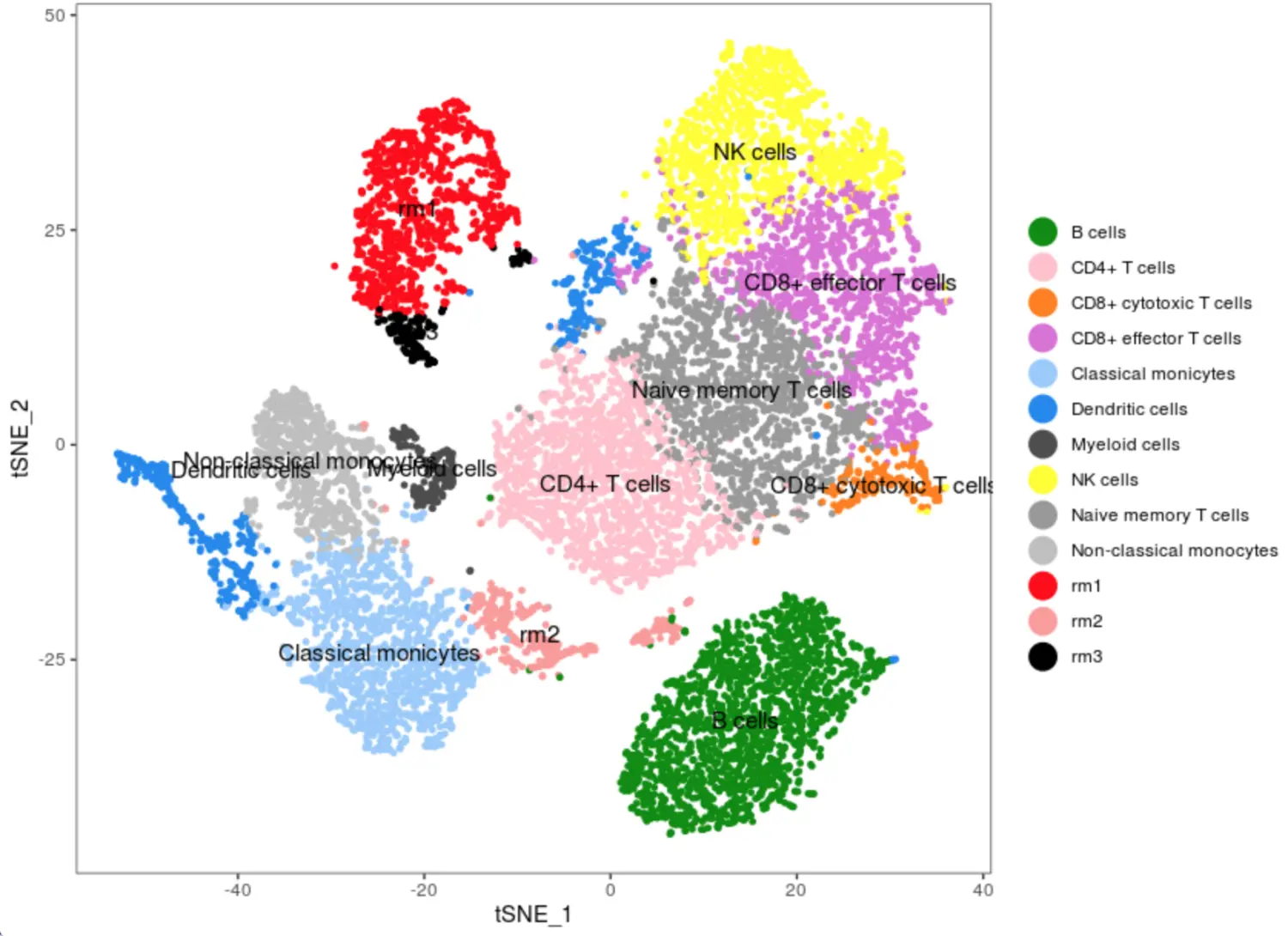
# 修改颜色顺序,思想就是:将现在的细胞名与表中的细胞名对应一下,然后这个顺序就是颜色出现的顺序
colP=colP[match(levels(as.factor(labels)),a[,2])]
TSNEPlot(PBMC, group.by = 'labels',
colors.use = colP,
do.label = T)

再按时间拆分分群结果
做出文章的这张图
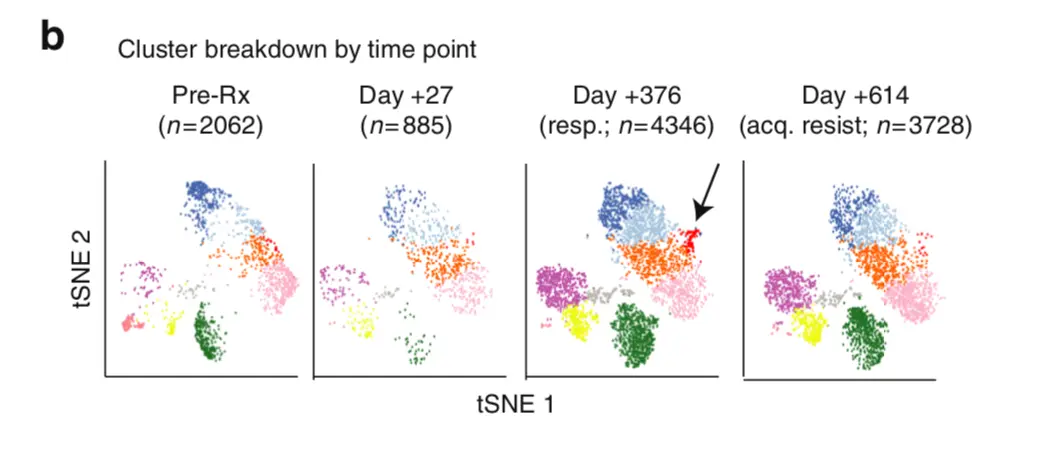
首先得到我们的四个时间点
> TimePoints = PBMC@meta.data$TimePoints
> table(TimePoints)
TimePoints
PBMC_ARD614 PBMC_EarlyD27 PBMC_Pre PBMC_RespD376
4516 1592 2082 4684
然后用一个函数SubsetData
举一个例子,绘制Pre时期的分群图
# SubsetData函数其实也是利用TRUE/FALSE取子集
PBMC_Pre = SubsetData(PBMC,TimePoints =='PBMC_Pre')
TSNEPlot(PBMC_Pre,
colors.use = c('green4', 'pink', '#FF7F00', 'orchid', '#99c9fb', 'dodgerblue2', 'grey30', 'yellow', 'grey60', 'grey', 'red', '#FB9A99', 'black'),
do.label = F)
# ggsave('PBMC_Pre_tSNE.pdf')