082-表观遗传初识~ChIP-seq
刘小泽开始于19.2.3
之前从未涉足这个领域,但确实是未来发展方向 还是老规矩,流程学习很快,重点在于为什么做以及做完怎么解读 先补充背景知识吧
背景知识
- 转录组即某个物种或特定细胞在某一功能状态下产生的所有RNA的总和 (哪些序列能够表达,何时何处表达, 转录活跃程度)
- 部分基因是能够转录=》染色质多为开放状态;其余基因抑制状态=》染色质状态较为紧密【基因开启、关闭:基因调控】
- 基因表达调控:信号转导、转录前(染色质构象和表观调控等)、转录调控、转录后调控(剪切、编辑、转运等)、翻译和翻译后调控【针对DNA序列:顺式元件Cis; 针对结合因子包括蛋白和非蛋白因子:反式作用因子Trans】
- 非编码区大量组织特异性调控序列:增强子enhancer、沉默子silencer、绝缘子insulator等+几个/几十/几百调控因子(转录因子、染色质调控蛋白、组蛋白、RNA分子)+三维结构
- 目前技术:
- 分离纯化多拷贝的基因组重复序列,如染色体端粒(telomere):locked nucleic acids (LNAs)和transcription activator-like (TAL)
- 常规技术:ChIP-seq和ChIA-PE依赖于单个调控因子或组蛋白修饰【不能纯化、分析单个调控序列所结合的多个调控因子及三维结构】
- 挑战:native状态下区分结合调控序列的特异性调控因子和细胞内大量的非特异性因子
文献综述一定要看
PPT大体印象;综述帮助理解;文献获取流程
PPT:
Basics of ChIP-Seq https://www.msi.umn.edu/sites/default/files/basics_chip_seq_0.pdf
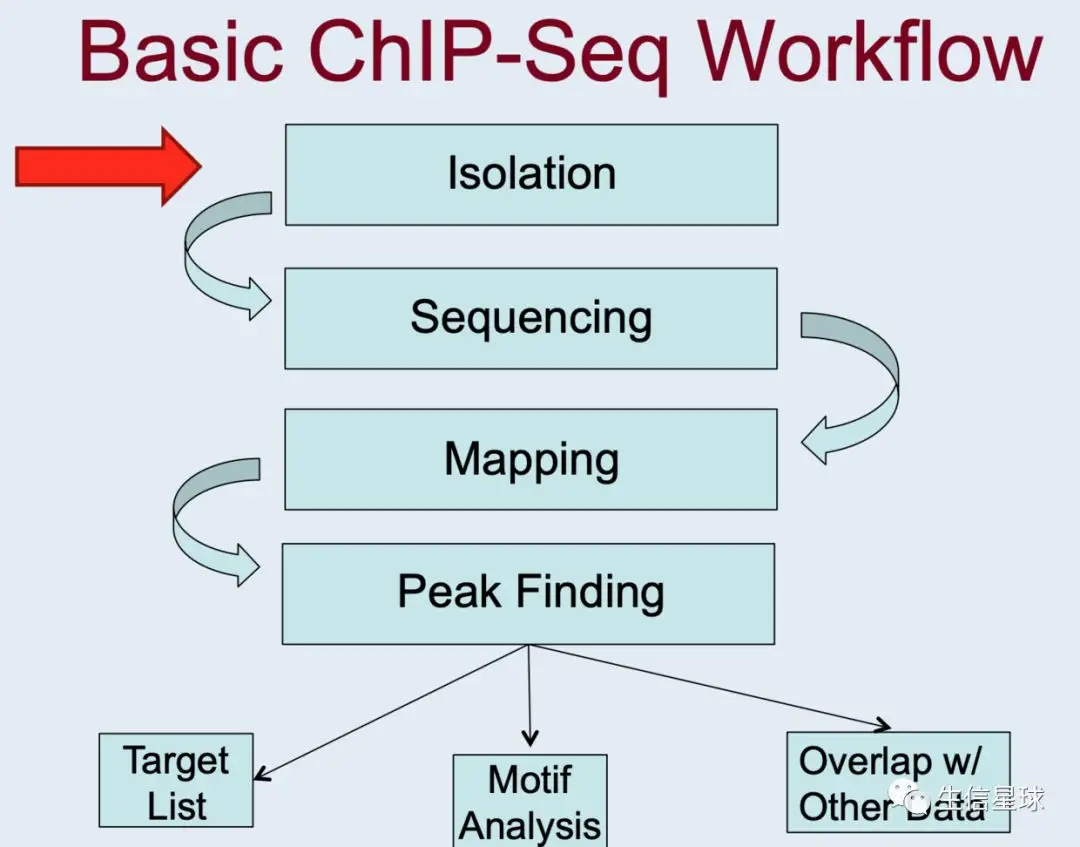
ChIP-seq Data Analysis https://slideplayer.com/slide/3385783/
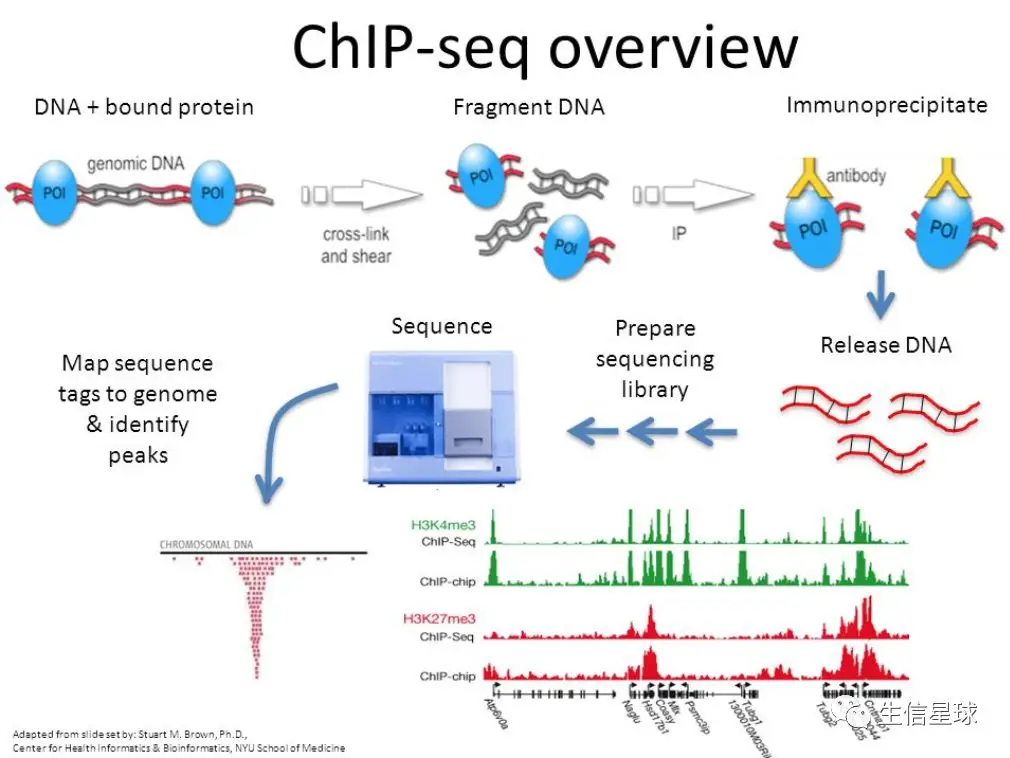
图中可以看到,基本原理是:特定时间点上用甲醛交联等方式“固定”细胞内所有DNA结合蛋白的活动,细胞内蛋白和DNA相互作用的关系被“截屏”了=》然后裂解细胞、断裂DNA(此时存在蛋白、与蛋白相连的DNA、游离的DNA)=》加上特定抗体取出蛋白及相连的DNA=》将与蛋白相连的DNA解离、纯化=》测序=》看到蛋白质抓取的片段们就是IGV中形成的峰,而游离的DNA们就没有峰
综述:
ChIP-seq guidelines and practices of the ENCODE and modENCODE consortia (2012) https://www.ncbi.nlm.nih.gov/pubmed/22955991
首先就介绍了ChIP的历史【染色质免疫沉淀(Chromatin immunoprecipitation ,ChIP)技术诞生很早,由Orlando等人创立于1997年,发表于2000年,先利用Microarray技术 ChIP-chip,后2007年利用DNA sequencing技术 ChIP-seq。虽然技术在进步,但都是要先通过免疫沉淀拉下来DNA】
它用来研究细胞内DNA与蛋白质相互作用,确定特定蛋白(如转录因子)是否结合特定基因组区域(如启动子或其它DNA结合位点),或确定基因组上与组蛋白修饰相关的特定位点
然后提到两个组织:ENCODE(http://encodeproject.org/ENCODE/ )和modENCODE(http://www.modencode.org/ for small genomes),他们在4个物种(果蝇、秀丽隐杆线虫、人、小鼠)的100多种细胞做了超过1000次ChIP-seq实验,发现了140多种不同的因子和组蛋白修饰 【与外显子测序不一样,ChIP-seq不是通过设计好的探针来捕获序列,而是通过特异的RNApoly酶、组蛋白、转录因子来捕获,哪里结合蛋白就捕获哪里。每做一次实验,换一个蛋白,所捕获的序列是不一样的】

文献:
Getting-Started-with-ChIP-Seq(2013)
http://epigenie.com/wp-content/uploads/2013/02/Getting-Started-with-ChIP-Seq.pdf
Key steps:
Experimental design sequencing platform; the number of sequence reads (standard 20-40M); biological replication
Controls for ChIP-seq experiments
Reference genome alignment of ChIP-seq reads
(mapping)
Background estimation
Peak finding
Quality control of ChIP-seq experiments
Differential binding analysis
Motif analysis