067-非编码miRNA的简单介绍
刘小泽写于18.12.15
本来不知道要写点什么,但是想起来自己一直接触的都是编码的RNA,对于大部分非编码RNA的还不了解,于是借着这次机会先稍微了解一下miRNA。感觉学习任何东西都是如此,先有个印象,不至于别人提到了它,自己却还一脸懵。学习不是一蹴而就,都是渐进式的过程,这次有个印象,下次说不定看文献看到就不陌生,再下次就开始看看里面有什么可以分析的内容。嗯,就是这样!
先看看什么是miRNA
miRNA是真核生物中的一类内源性的具有调节基因表达功能的非编码RNA,长度为20-25nt(这里注意nt和bp的区别: 我自己之前就总是搞混,bp就是base pair,成对的碱基,大多出现在DNA中;nt为核苷酸的简写,对于单链的RNA来讲,就是用nt来描述啦)。成熟的miRNA是由较长的初级转录产物经过一系列的核酸酶剪切加工形成。至于作用嘛,就是合成后再组装进RNA诱导的沉默复合体(RNA-Induced Silencing Complex, RISC),通过碱基互补配对的方式识别靶mRNA【根据互补程度的不同指导RISC降解靶RNA或者阻遏靶RNA的翻译】(来自:DOI: 10.1038/ncb0309-228 )
一般来讲主要有这几个特点:
- 目前超过80%的miRNA长度在21-23nt之间
- 含有能形成茎环结构的前体【其中动物的前体大小在60-80nt之间,植物变化范围比较大,在几十到几百nt】
- 成熟的miRNA在5‘有一个磷酸基团,3’有羟基,可以和上游或者下游的序列不完全配对形成茎环结构
- 在基因组中有多种存在形式,包括单拷贝、多拷贝、基因簇等,位置大多在基因间区(说明它们的转录是独立于其他基因的,有自己的一套调控机制)
- 大多数miRNA是从前体的一条臂上加工而来的,大部分前体的两条臂可以分别产生一个miRNA
- 生物发育的不同阶段,或者不同组织中,有不同的miRNA表达水平,可能miRNA参与了发育和行为变化的调控
miRNA的合成与功效?
纯粹的生物背景知识,可以从任何生物课本上学到
miRNA是1993年由哈佛的Lee等人在研究C.elegans 发现的
miRNA一般由RNA Pol II 转录,转录的最初产物是具有帽子结构和多聚核苷酸尾巴(poly-A)的pri-miRNA(300-10Kbp),再加工成具有茎环结构的前体pre-miRNA(70-90bp),再在Dicer酶切作用下将pre-miRNA的5‘和3’剪切掉,形成成熟miRNA。
对于哺乳动物来讲,pri-miRNA的处理是在核内,依靠microprocessor复合物进行的【复合物包括了RNase III enzyme Drosha, DGCR8(DiGeorge critical region-8)以及一个双链RNA结合蛋白组成】
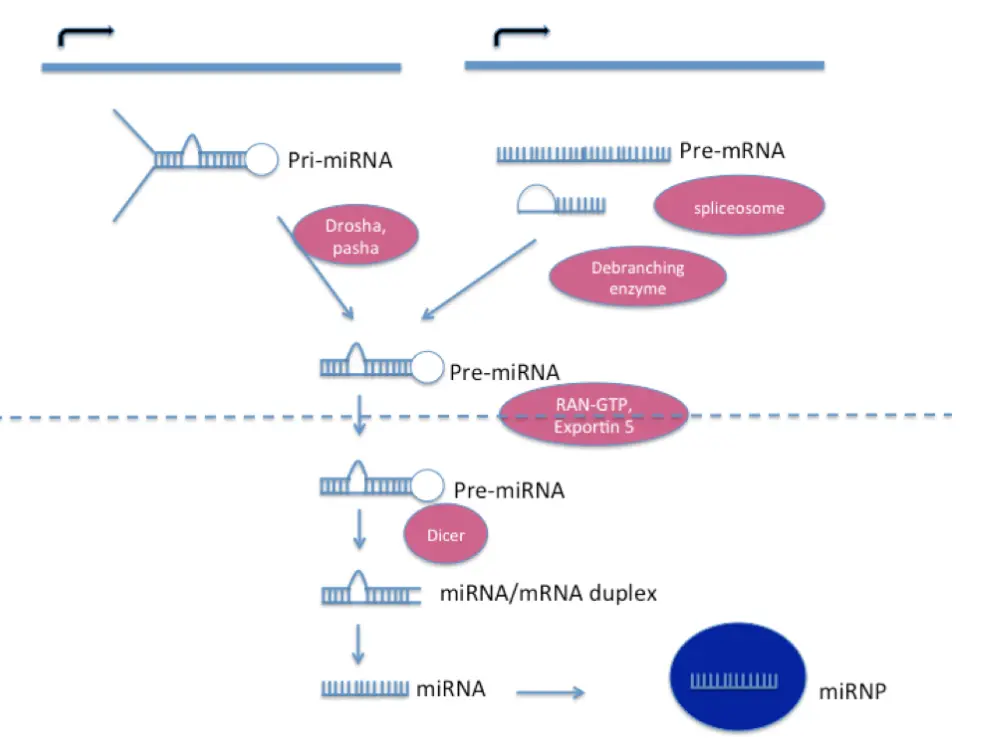
miRNA一般在转录后调控方向研究较多,比如miRNA的5’端与mRNA的3‘UTR区域结合(即seed区域)【至少7个连续核苷酸完全结合配对时就会降解mRNA;部分配对会产生负调控,抑制mRNA翻译】 ,于是衍生出了靶基因预测的研究方向
关于靶基因预测
序列互补性
种子序列(seed)位于miRNA的5‘端第2-8nt,与靶基因的3’UTR是否可以形成配对,这个是判断miRNA靶基因的关键
保守性
miRNA的结合位点在多个物种之间如果有保守性,那么这个位点更可能是靶位点
热动力学
主要考察miRNA:target对形成的自由能,自由能越低,可能性越大
位点可结合性
考虑mRNA二级结构的影响
UTR碱基分布
miRNA结合位点在UTR区域的位置和相应位置的碱基分布
针对以上几点,开发许多的预测软件:
- miRanda:最早的miRNA靶基因预测软件,对3‘UTR的筛选依据主要是序列匹配、miRNA与mRNA双链的热稳定性以及靶位点的跨物种保守性。选取每条miRNA相对的3’UTR中排名前10的基因,作为候选靶基因。如果多个miRNA对应一个靶位点,就根据贪婪算法选择得分最高并且自由能最低的那对
- TargetScan:根据靶基因跨物种保守与miRNA-靶基因二聚体热力学特征开发。需要种子序列(2-8nt)和mRNA的3‘UTR完全互补,并且从种子序列向两侧延伸,直到遇到不能配对的碱基(允许G-U配对)
- miTarget:基于对miRNA和靶基因的二聚体结构、热力学特性和miRNA与靶基因作用的碱基位置等参数,利用机器学习支持向量机,可以选择好的数据集,提取特异性高的参数,而不用考虑靶基因的跨物种保守性
- PicTar:根据种子序列在靶位点识别及转录后调控的关键作用,将seed分为完全匹配的seed和不完全匹配的,同时对两类seed的miRNA与靶基因二聚体结合进行限制,降低假阳性率
- RNAhybrid:根据miRNA和靶基因二聚体二级结构开发,本质是预测RNA二级结构的软件。它根据miRNA和靶基因间的结合探测最好的靶位点,不需要考虑靶基因的物种保守性