044-生物数据库ID,让我深入了解你
刘小泽写于18.10.15 内容很多,资料就准备消化了一上午
基因的身份实在太多,因为每个数据库都想有自己的独到之处,因此我们有必要对这么多基因的ID进行分类整理,分清主次才能从容应对
不要被吓到哦
这是一张生物数据库网络图,上百种ID乍一看杂乱无章,但是仔细一看,主要靠的就是灰色背景的这三大数据库:NCBI的Entrez、Ensembl、Uniprot;蓝色背景数据库是目前比较主流的数据库,如RefSeq、GenBank、dbSNP、GO、KEGG等,与三大数据库有数据交换;没有背景的就是主流数据库产生的各种ID,包括了我们日常经常使用的HGNC ID、PubmedID、Pfam ID、UCSC ID、RefSeq ID、Enzyme ID、GO term、KEGG patheway等,另外还有一些物种专属数据库ID,比如WormBase ID、TAIR ID、FlyBase ID等
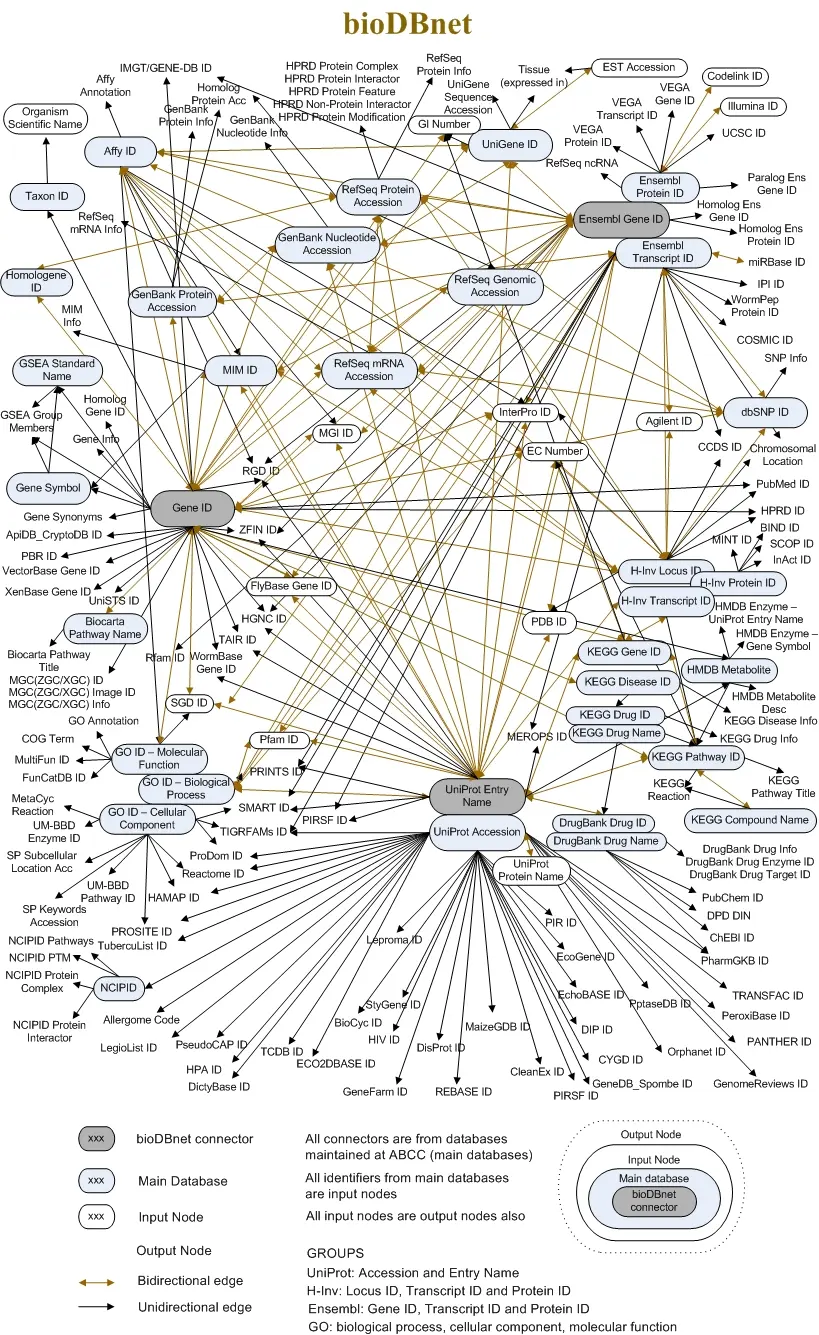
下面就层层递进来介绍这些主要的数据库 先了解数据库,再了解ID,效果更好哦
第一级 三大数据库
1 NCBI家的Entrez
1.1 历史背景
先搞清楚:NCBI是一个组织,Entrez是它的搜索系统
NCBI是美国国立生物技术信息中心(National Center for BiotechnologyInformation),由美国国立卫生研究院(NIH)于1988年创办,目的是为生物学家提供一个信息储存和处理的系统。它建立了自己的核酸序列数据库GenBank,与日本的DDBJ(1986年创办)、欧洲分子生物学实验室的EMBL数据库(1982年创办)等联合存储。东西一样但命名各异。
如果要在NCBI中检索信息(基因序列、基因组、基因型、基因表达、分子结构、蛋白质组学、文献等等),就需要用到Entrez的综合检索系统。 Entrez的第一个版本由NCBI于1991年在CD -ROM上发布,当时核酸序列来自GenBank和Protein Data Bank(PDB)数据库,蛋白序列来自GenBank、Protein Information Resource (PIR)、SWISS-PROT、PDB以及Protein Research Foundation (PRF)数据库,还从MEDLINE数据库(现在是PubMed)整合了文献摘要。
1.2 内容
基因序列数据库
Gene:基因序列注释+检索,目前共有61118个人类的记录,68389个小鼠的记录(含有功能基因、假基因、预测基因等),并且每天更新。
大家都曾年轻过,如今风华正茂的Gene数据库曾经叫做LocusLink,当时只含有人类的数据,并且也只有少于9000条记录,它的好朋友也只有GenBank、Unigene、dbSNP、OMIM,2003年正式更名为Gene。
Unigene:属于GenBank一部分,包含转录本序列,包括EST序列以及非冗余序列。Unigene的意思就是潜在的基因
HomoloGene:在完全测序的真核生物基因组中检索同源基因,以及上下游序列
Reference Sequences(RefSeq):注释过的非冗余转录体、蛋白质和基因组序列数据库(ftp://ftp.ncbi.nih.gov/refseq/release/)
基因组数据库
- Genome:真核生物完整基因组组装数据及注释数据+动植物、真菌染色体信息(ftp://ftp.ncbi.nih.gov/genomes/)
- Trace archives:一代测序数据
- Short Read Archive(SRA):二代测序数据
基因型与表型数据库
- dbGaP(www.ncbi.nlm.nih.gov/sites/entrez?db=gap):数据来自NIH提供的GWAS全基因组关联分析
- dbSNP:单核苷酸多态性信息、种群特异性等位基因频率、个体基因型
- dbMHC:组织相容性复合体(MHC)等位基因的变异信息,与器官移植、感染病敏感性有关
- dbLRC:白细胞受体复合物(LRC)等位基因(如:KIR基因)
- dbRBC:红细胞抗原或血型有关基因
- OMIM:人类遗传病数据库,包括遗传病详细的描述、基因名称、遗传方式、基因定位、基因多态性、文献,由约翰霍普金斯大学维护
基因表达数据库
- GEO(Gene Expression Omnibus):芯片和二代测序实验数据,包括基因表达、基因组拷贝数变异、基因组-蛋白互作、甲基化。有原始数据和处理过的数据
- GENSAT:小鼠中枢神经系统基因表达谱
- Entrez Probe:探针试剂信息、销售厂家信息、探针有效性,涵盖用于检测基因沉默、基因表达、SNP、基因分型、基因测序等的探针
分子结构、蛋白数据库
- MMDB:蛋白质结构域注释、PDB异质基因、保守结构域、结构邻域信息
小分子数据库
- PubChem附属的三个数据库:PCSubstance、PCCompound和PCBioAssay,建立了基因组水平的生物大分子与细胞代谢水平的小分子之间联系
2 Ensembl
2.1 背景知识
Ensembl项目始于1999年,也就是人类基因组草案完成前几年,由英国的Sanger研究所以及欧洲生物信息学研究所(EMBI-EBI)联合共同协作开发。发展的起因是手动注释30亿个碱基序列对数据的时效性考验,于是Ensembl想要自动注释基因组,并将注释和其他可用的生物数据相结合。2000年7月上线网站,侧重于脊椎动物的基因组数据,但后来逐步丰富了其他生物如线虫,酵母,拟南芥和水稻等。
大多数物种需要三到六个月才能使用Ensembl自动注释系统进行注释,这个过程是这样的:整合Uniprot KB的蛋白信息与NCBI RefSeq里面的mRNA信息,再加上人工注释【人工注释在VEGA和Havana数据中可以找到】
关于自动注释和人工注释:自动注释很快,但是它是基于概率的过程,不一定完全准确;人工成本就比较高,但准确度也高

2.2 内容
FTP下载内容:https://asia.ensembl.org/info/data/ftp/index.html
FASTA:Ensembl基因的FASTA序列数据库,转录本和蛋白质模型预测
GTF/GFF3: 编码和非编码基因的注释
需要注意的是:基因组和GTF文件中染色体名字都没有添加
chrVCF:变异数据
BED:Browser Extensible Data,为genome browser而生
3 Uniprot
3.1 背景知识
它是Universal Protein的简写,整合了Swiss-Prot、TrEMBL和PIR-PSD三大数据库,含有最丰富的蛋白质数据。通过Uniprot 数据库,我们可以了解编码蛋白的基因,对应的蛋白名称, 序列,以及GO注释(GO ID、GO的描述信息、分类这些信息是经常需要用的)
SWISS-PROT
注释过的蛋白质序列数据库,由欧洲生物信息学研究所(EBI)维护。只接受直接测序获得的蛋白质序列。每个蛋白质条目包括了序列、文献、分类、注释,注释又包括:功能、转录后修饰、特殊位点、二级和四级结构、相似性大小、序列变异、序列残缺与疾病联系
SWISS-PROT的网址:http://www.ebi.ac.uk/swissprot/
PIR-PSD
国际蛋白质序列数据库(PSD)是由蛋白质信息资源(PIR)、慕尼黑蛋白质序列信息中心(MIPS)和日本国际蛋白质序列数据库(JIPID)共同维护的国际上最大的公共蛋白质序列数据库。特点是:全面、注释、非冗余,基本都按基因家族进行了分类,一般以上进行了蛋白质超家族分类。
PIR和PSD的网址:http://pir.georgetown.edu/ 数据库下载地址:ftp://nbrfa.georgetown.edu/pir/
另外还有:
PROSITE
包含有显著生物学意义的蛋白质位点和序列模式,可以鉴别一个未知功能的蛋白质序列应该属于哪一个蛋白质家族。序列模式包括:酶的催化位点、配体结合位点、与金属离子结合的残基、二硫键的半胱氨酸、与小分子或其它蛋白质结合的区域
PROSITE的网址:http://www.expasy.ch/prosite/
PDB
由美国Brookhaven国家实验室建立的唯一的生物大分子结构数据档案库,由结构生物信息学研究合作组织(RCSB)负责维护。它主要搜集X光晶体衍射和核磁共振(NMR)的数据,使用Rasmol等软件可以按PDB文件显示生物大分子的三维结构
RCSB的PDB数据库网址:http://www.rcsb.org/pdb/
COG
蛋白质直系同源簇数据库,依据系统进化关系分类建立。用于预测单个蛋白质以及整个基因组中蛋白质的功能
COG库的网址:http://www.ncbi.nlm.nih.gov/COG 数据库:ftp://ncbi.nlm.nih.gov/pub/COG
SCOP
蛋白质结构分类数据库,主要描述已知的蛋白质结构之间的关系,包含近缘的家族、远缘的超家族、空间几何结构折叠子(fold)等
SCOP的网址:http://scop.mrc-lmb.cam.ac.uk/scop/
3.2 内容
这些数据来源是:基因组测序项目完成后获得的蛋白质序列
- UniProtKB/Swiss-Prot:手工注释的、非冗余高质量的数据集
- UniProtKB/TrEMBL:自动注释的未经校验的计算结果,为了弥补校验基因组数据人力不足的缺陷。可以注释所有可用的蛋白序列,包括三大核酸数据中注释的编码序列以及PDB的序列,另外还有Refeq、CCDS、Ensembl的基因预测序列 前两个合称UniProtKB(即UniProt Knowledge Base)
- UniParc(UniProt Archive):包含所有公开的蛋白质序列【没有注释】,并且无论同一个蛋白质序列出现在多少个数据库,UniParc中只有一个记录。并且不管是否为同一物种的序列,只要序列相同就被合并为一条,还有唯一的UPI编号
第二级 主流数据库
基础数据库
HGNC
官网 https://www.genenames.org/ 整个数据地址在:ftp://ftp.ebi.ac.uk/pub/databases/genenames/new/tsv/hgnc_complete_set.txt
我们常说的“TP53”、“BRCA1”等就是官方认证的基因名
早在20世纪60年代研究者就已经认识到人类遗传学命名的问题,1979年在爱丁堡人类基因组会议(HGM)上提出了人类基因命名的完整指南。HGNC由美国国家人类基因组研究所(NHGRI)和 Wellcome Trust(英国)共同资助,其中的每个基因只有一个批准的基因symbol。2007年9月,HGNC迁至欧洲生物信息学研究所(EBI),加入PANDA(蛋白质和核苷酸数据库)小组
目前已经批准了近33000个symbol,绝大部分是蛋白编码基因,当然也包括假基因,非编码RNA,表型和基因组特征的symbol
这里列出了HGNC的统计数据http://www.genenames.org/cgi-bin/statistics
目前总共有41506个认证的基因symbol 编码蛋白的有19198个(大多有GO注释) 非编码RNA共7375个,包括lnc、micro、smallRNA等 假基因13188个;免疫相关、病毒相关基因有1174个
HGNC会根据不同类型的基因,提供不同的参考数据库:
- 蛋白编码基因:提供uniprot的ID,例如“BRCA1”编码的蛋白质在uniprot数据库中ID是P38398;
- microRNA基因:提供miRBase数据库的信息;
- LncRNA基因:提供LncRNAdb的信息
- 当然还有其他许多类型的基因也有各自的数据库链接
GeneCards
它真的很好的表达了“不生产数据,只运输数据”的理念,绝对的低调奢华有内涵。
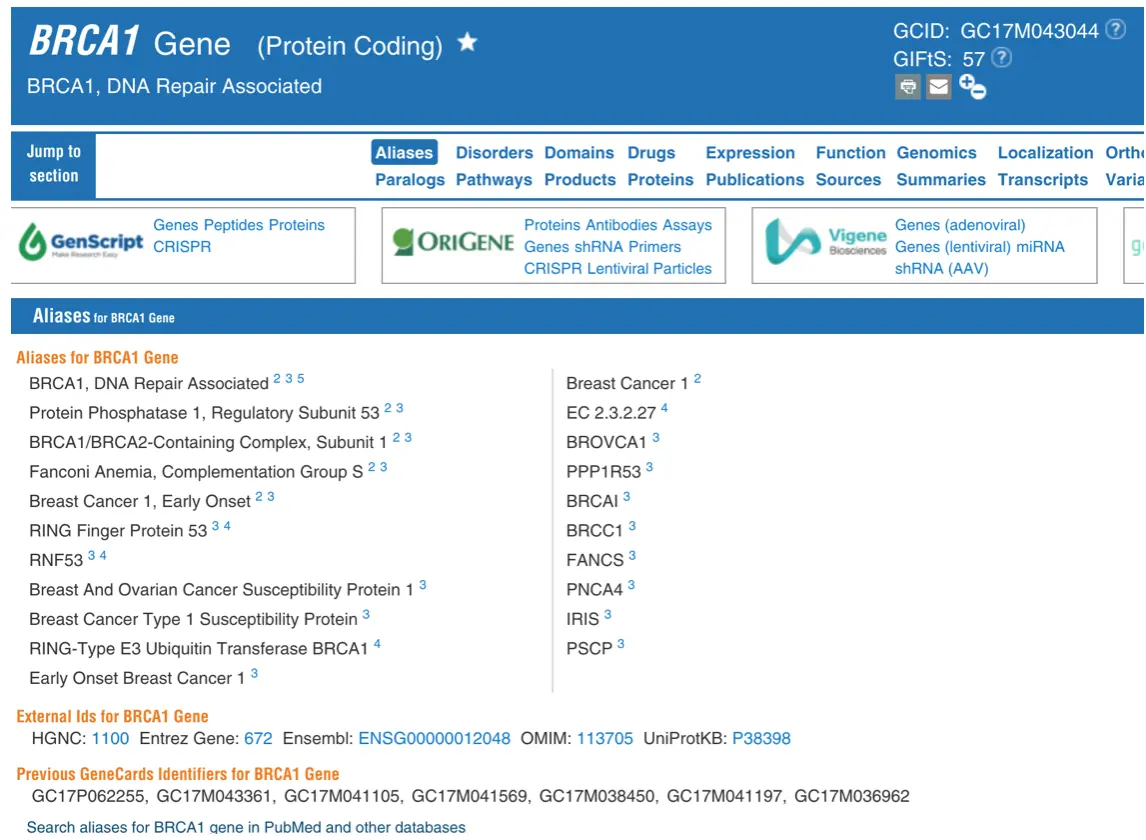
它是由Crown HumanGenome Center创办并更新维护的人类基因数据库,整合了125个左右的数据库资料,涵盖了基因组、转录组、蛋白组、临床、功能信息等
从上往下,往细了说就是: 1 全称、属性(编码蛋白、基因簇、假基因等); 2 通用名、其他数据库ID号; 3 来自多个数据库对主要功能描述; 4 基因组相关信息:位置、DNA序列、转录因子、调控元件、表观遗传、甲基化; 5 蛋白相关:亚型、大小、二三级结构、翻译后修饰、蛋白抗体; 6 蛋白结构域及家族信息; 7 基因功能:GO、表型、互作miRNA、动物模型、siRNA、基因克隆、原位杂交 8 通路及互作蛋白; 9 相关药物与化合物; 10 转录本信息; 11 表达量:芯片、RNA、SAGE、共表达: 12 不同物种保守性 13 旁系同源基因、假基因 14 SNP与疾病相关 15 相关疾病 16 相关文献 17 外部其他100多个数据库链接
GenBank
官网 https://www.ncbi.nlm.nih.gov/genbank/ 文件下载:ftp://ftp.ncbi.nlm.nih.gov/genbank
它是NIH(National Institute of Health)附属的注释核酸数据库,两个月一次更新。刚开始1982年release3包含606条序列,共680338个碱基。现在已经到了227版。来源也扩充到了260000+物种,其中最多的是人类(约13%)
主要包括:
WGS(Whole Genome Shotgun):未注释的全基因组测序序列数据库
TSA(Transcriptome Shotgun Assembly):转录组测序组装序列数据库
TLS(Targeted Locus Study):特定位点研究
【后来这三个为了避免重复统计,都开始各自单独统计】
一般我们下载核酸数据,都会发现有两个选项:FASTA和GenBank。FASTA只记录基因的精简信息,用ATCG表示出核苷酸序列就好了;而GenBank可以表示比较完整的基因序列信息

RefSeq
参考序列数据库,包含具有生物意义的非冗余基因、转录本和蛋白质序列,经过NCBI及其他组织的校正,使用HGNC指定的标准名。这里的序列,就是可以用来对照的,其中包含染色体、基因组、RNA、蛋白等。
RefSeq与GenBank的不同就是:
- GenBank开放,每个基因都有许多序列,研究者、公司都可以自己提交序列,而且它每天还和EMBL、DDBJ交换数据,因此数据冗余度高,准确度可能要低一些;有时会产生别名
- RefSeq是按照人类每个位点挑一个代表序列构建起来的,并且是NCBI精挑细选后得到的,一般可信度较高;全部使用官方基因符号
每个条目的后面都会有状态信息:PROVISIONAL REVIEWED表示已被人工审核;PREDICTED表示没有经过审核;MODEL表示是由NCBI自动提交的,未经审核;INFERRED表示由序列预测得到的,没有实验验证,VALIDATED初步审查过,但还没最终审查
NR与NT
下载地址 ftp://ftp.ncbi.nih.gov/blast/db,解压缩后构建索引文件即可
NR:非冗余蛋白库 Non-Redundant Protein Sequence Database,包括所有的GenBank+EMBL+DDBJ+PDB中的非冗余蛋白序列。它以核酸序列为基础进行交叉索引,将核酸与蛋白质联系起来。对于已知的或可能的编码序列,NR记录中都给出了相应的氨基酸序列(由读码框推断)。
NT:核酸序列数据库(Nucleotide Sequence Database),是NR库的子集
dbSNP
官网 http://www.ncbi.nlm.nih.gov/SNP/ 数据库 https://ftp.ncbi.nih.gov/snp/
SNP是单核苷酸多态性,就是DNA序列中一个特定位点出现两个或者多个A、T、C、G的改变,这种多态性占所有已知多态性的90%以上。人类基因组中平均500-1000个碱基对就有1个SNP,目前发现了大概400万个SNPs。
单核苷酸多态性数据库是由NCBI与人类基因组研究所(National Human Genome Research Institute)合作建立的,起初的目的是对GenBank提供补充,后来并入了Entrez系统,可以直接查询。主要记录了单碱基替换、插入、缺失的情况。
数据库中最有用的目录是:
- organisms:包含SNP数据的生物体,命名方式是:
通用名_分类ID,例如:monkey_78449 - database:包含模式、数据、创建表格与索引的SQL语句(这里要知道SNP数据库结构是“中心辐射状”,中心是dbSNPmain表格,辐射的是具体生物体的数据库)
- specs: 目录包含重要的文件的格式,内容及其基本介绍
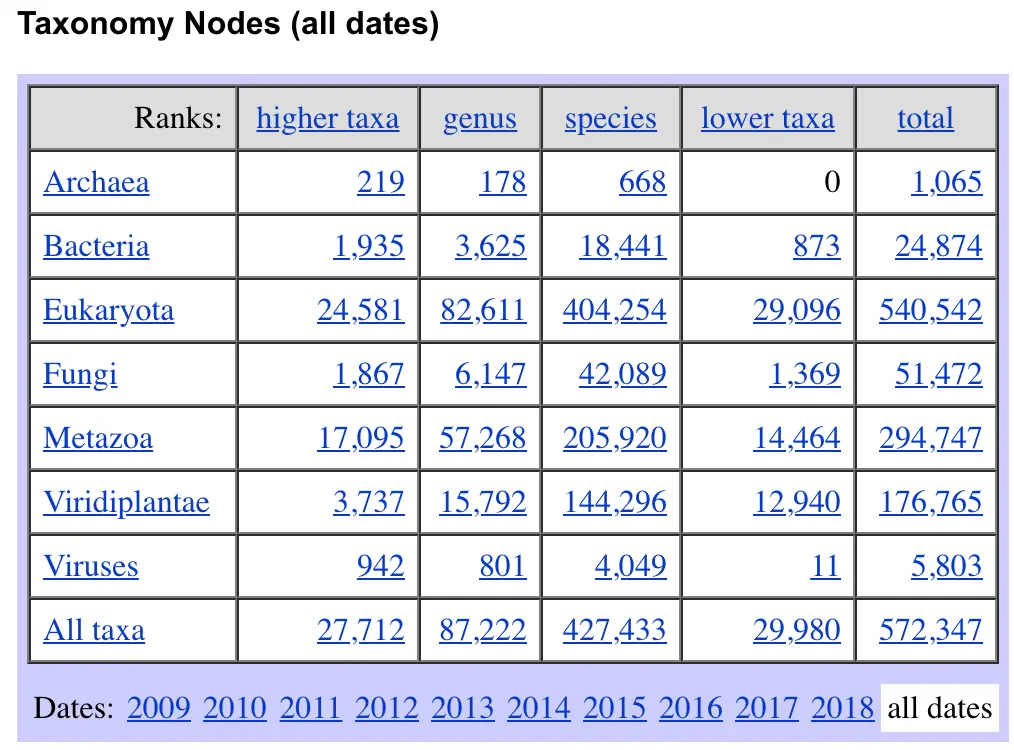
Taxonomy
分类数据库
重要的三个文件:
gi_taxid.nucl.dmp.gz(NT记录ID号与taxid对应关系)gi_taxid.prot.dmp.gz(NR记录ID号与taxid对应关系)taxdump.tar.gz(其中包含names.dmp和nodes.dmp) names.dmp有四列,|分隔,包括记录号tax_id、物种名称name_txt、unique name、name class nodes.dmp有13列,|分隔;物种分类注释时需要tax_id(Taxonomy记录号),parent tax_id(上一层分类级别的tax_id)和rank(该tax_id所处的分类层级)
专用数据库
GO
这是一个强调基因产物功能的数据库,并非基因序列或基因产物数据库。
它建立的初衷是为了减少生物学定义的混乱,为了对各种数据库中基因产物功能的描述打造成一致的结果。项目最初是由1988年对三个模式生物数据库的整合开始:: FlyBase (果蝇数据库Drosophila),t Saccharomyces Genome Database (酵母基因组数据库SGD) and the Mouse Genome Database (小鼠基因组数据库MGD),现在已包含数十个动物、植物、微生物的数据库。它由国家人类基因组研究所 (NHGRI)以及欧盟RTD项目赞助,免费使用但必须引用基因本体联合会。
它的组织形式就是三级结构的标准语言,也叫做本体论(Ontology):
- 分子功能本体论 基因产物个体的功能,如与碳水化合物结合或ATP水解酶活性等
- 生物学途径本体论 分子功能的有序组合,达成更广的生物功能,如有丝分裂或嘌呤代谢等
- 细胞组件本体论 亚细胞结构、位置和大分子复合物,如核仁、端粒和识别起始的复合物等
GO中的术语是如何与相对应的基因产物联系的呢?这就要靠参与合作的数据库来完成,它们使用GO的定义方法,对基因产物(基因编码的RNA或蛋白质)标注并提供理论支撑,并给出基因产物和GO术语联系的数据库(当然术语不能太长,能粗略估计就好)。这样可以帮助判断蛋白结构域功能,预测某种疾病相关基因,分析发育过程中共表达基因,找到某些异常表达基因的功能相关性等。
GO推荐的注释是针对基因产物的而不是基因的,因为一个基因可能编码多个具有很不相同性质的产物,这样有助于更好地阐明基因产物和GO术语之间的联系。
需要注意的是,GO注释都是反映的正常情况下的基因产物的功能,不包括突变或者病变的情况
GO数据有三种格式:flat(每日更新)、XML(每月更新)和MySQL(每月更新)
KEGG
全称是:Kyoto Encyclopedia of Genes and Genomes,大概可以分为系统信息、基因组信息、化学信息和健康信息四大类,共包含了17个主要的数据库,是生物体代谢网络分析重要的工具。其中最核心的是KEGG Pathway数据库,又分为3个层级:
- 第一层级:生物代谢通路分为7个大类,新陈代谢、遗传信息加工、环境信息加工、细胞过程、生物体系统、人类疾病、药物开发;
- 第二层级:将第一层级中的7个类别进一步细化;
- 第三层级:直接对应KEGG 的pathway,每一个pathway都标示参与该过程的基因
基于KEGG注释结果,可以快速寻找某类功能的基因,同时构建代谢通路图;并且将各步反应催化的酶的信息进行标注,还加上了氨基酸序列信息,支持链接到PDB
GEO数据库
官网 https://www.ncbi.nlm.nih.gov/geo/ 快速查找GEO:https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE17708 (只要将17708换成其他数字即可抵达)
随着测序技术越来越先进,数据产量也越来越大,NCBI为了整合零散的基因表达数据,开始了基因表达汇编计划,建立了最大最全面的基因表达数据仓库(Gene Expression Omnibus)。
数据来源可以是芯片数据、SAGE、高通量测序mRNA、lncRNA等
数据存放:四种类型GSE、GSM、GPL、GDS
- GSE将整个项目的一系列样本和平台联系起来,比如GSE17708(都是GSE+数字)GSE=GPL+GSM
- GSM对应一个样本的数据,只能对应一个平台,表示每个样本操作环境
- GPL就是平台信息,包含微阵列或者测序平台简要描述(GPL+数字)
- GDS就是同一个平台的数据集:如微阵列进行的表达谱分析或则非编码RNA分析;微阵列进行的ChIP分析、甲基化分析;高通量测序;SNP阵列;蛋白质阵列
数据下载:利用R包GEOquery的getGEO函数
OMIM数据库
20世纪60年代,Dr. VictorA. McKusick发起建立人类孟德尔遗传的知识库,希望通过书籍出版的方式传播孟德尔遗传的表型性状和基因的知识。1995年,OMIM由国家生物技术信息中心NCBI为web开发
它的全称是:Online Mendelian Inheritance in Man(在线人类孟德尔遗传),截止2018年10月13日共收录了24705个词条,主要包括15981个基因词条和5337个已知分子机制的表型词条,包含了有关所有已知孟德尔病症
主要根据OMIM号左上角的符号区分:
*表示基因,如*113705+表示基因与表型组合,如+133430#表示已知分子机制的表型,如#114480%表示未知分子机制表型,如%607086
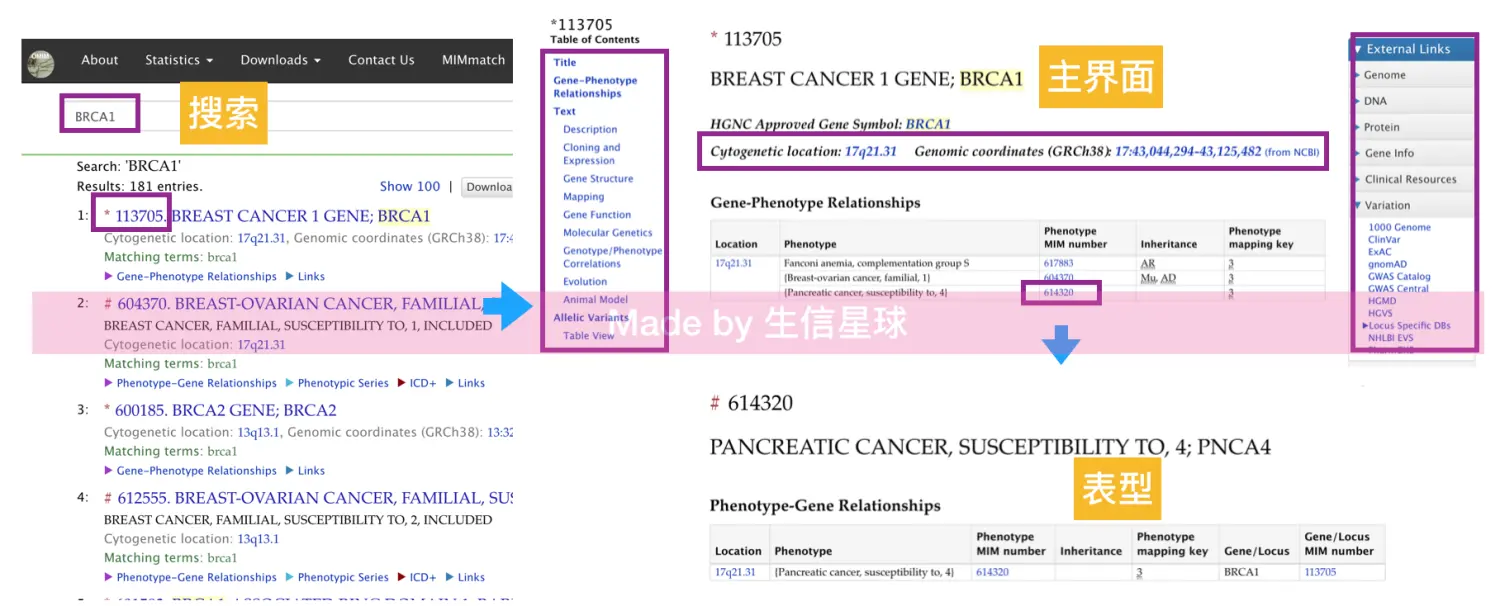
输入一个基因(不区分大小写),就会检索相应的词条:
- 中心部位是全称、HUGO symbol(也就是官方命名,不是别名)、在染色体上位置、物理位置
- 左侧是描述信息、克隆表达、基因结构、基因功能等;
- 右侧是有关基因的外部链接;
- 中心区域还包括表型检索号,点击就会得到相应表型信息。左侧是临床特征、遗传模式、分子机制等;右侧是有关表型的链接
ClinVar
官网 https://www.ncbi.nlm.nih.gov/clinvar/ 数据库 ftp://ftp.ncbi.nlm.nih.gov/pub/clinvar/
NCBI维护的与疾病相关的人类基因组变异数据库,整合了dbSNP、dbVar、Pubmed、OMIM等多个数据库,整理出变异、临床表型、实验数据、功能注释的信息,并且经过专家评审。因此ClinVar中有的,dsSNP中也有
系统采用星标打分,评估某个特定突变在疾病中的注释。四星等级最高,表示这个突变经过了大多数专家的认可;而没有星的,一般就是没有实验验证,不能提供理论支持。
clinvar的注释,可以寻找出对应的基因变异信息,发生频率,表型,临床意义,评审状态以及染色体位置等。
第三级 ID
之前在“一个萝卜一个坑https://www.jianshu.com/p/8ad714617fca”中提到了一些,比如Entrez ID、Ensembl ID等,这次再进行一个补充
| BRCA1基因ID示例 | ID数据库来源 |
|---|---|
| BRCA1 | HGNC gene symbol |
| 1100 | HGNC ID |
| 672 | Entrez ID, NCBI |
| ENSG00000012048 | Ensembl |
| NC_000017.11 | RefSeq, NCBI |
| P38398 | UniProt, accession number |
| BRCA1_HUMAN | UniProtKB/Swiss-Prot, entry name |
| U14680.1 | GenBank |
| 555931 | GenBank GI |
Gene Symbol 命名
【最容易辨认】全部由大写字母(或加数字)构成;当然也有HGNC自家的纯数字ID
Entrez ID 命名
【最广泛使用】纯数字表示,并且【再次提醒:不同物种的基因ID是不同的!】
多种工具支持Entrez ID与其他ID相互转变,除了clusterProfiler,还有ensembl下属的biomart
Emsembl ID命名
根据不同物种设置的前缀 + 数据类型(基因/蛋白质)+ 一系列的数字(例如版本号就以小数点体现)
2-3个月更新一次,目前是version 94。可以保证大多数情况下每次更新后名称保持稳定; 但是如果是数据自身发生变动(比如转录本更改),虽然主要ID信息不变,但是会增加小数,代表版本号。只有当变动特别大,比如重新组装基因组,才会更改整体名称。可以通过Ensembl的history ID来查看历史版本
| Ensembl物种前缀 | 名称 |
|---|---|
| ENS | Homo sapiens (Human) |
| ENSMUS | Mus musculus (Mouse) |
| ENSRNO | Rattus nornegicus (Rat) |
| ENSCAF | Canis lupus familiaris (Dog) |
| ENSDAR | Danio rerio (Zebrafish) |
| FB | Drosophila melanogaster (Fruitfly) |
| ENSXET | Xenopus tropicalis (Xenopus) |
| Ensembl类型前缀 | 名称 |
|---|---|
| E | exon |
| G | gene |
| P | protein |
| T | transcript |
| FM | Ensembl protein |
| R | regulatory feature |
| GT | gene tree |
RefSeq ID 命名
模式是:两个大写字母+一个下划线+大于6个数字
| RefSeq前缀 | 类型 | 说明 |
|---|---|---|
| AC_ | Genomic | Complete, alternate assembly |
| NC_ | Genomic | Complete, reference assembly |
| NG_ | Genomic | Incomplete genomic region |
| NT_ | Genomic | Contig or scaffold, clone-based or WGS |
| NW_ | Genomic | Contig or scaffold, primarily WGS |
| NS_ | Genomic | Environmental sequence |
| NZ_ | Genomic | Unfinished WGS |
| NR_ | RNA | |
| NM_ | mRNA | |
| XM_ | mRNA | Predicted model |
| XR_ | RNA | Predicted model |
| AP_ | Protein | Annotated on alternate assembly |
| NP_ | Protein | Associated with an NM_ or NC_ accession |
| YP_ | Protein | |
| XP_ | Protein | Associated with XM_ |
| ZP_ | Protein | Associated with NZ_ |
GenBank ID
【逐渐被refseq替代】AC号或者GI号 AC号:一般是一个大写字母加5个以上的数字,或者两个大写字母加上6个数字的组合 gi(GeneInfo identifier)号:只是一串数字,与AC号没关系
当数据发生变动,AC号主体不动,像ensembl一样更改小数点后的数字;而gi号会全部变动
UniprotKB ID
每个收录的数据都有一个唯一的Entry name或者Accession number。
Entry name最多支持11个字符的字母+数字的格式。命名方式是:“X_Y” 的形式,X是最多五个便于记忆的蛋白质编号,Y是最多五个便于记忆的物种编号,例如:
蛋白质/基因缩写+下划线+物种编码(属名前3个字母+种名前2个字母)
Accession number是最稳定的ID形式,并且唯一
| 蛋白编号 | 蛋白名称 | 基因名称 |
|---|---|---|
| CAD17 | cadherin-17 | CDH17 |
| HBA | Hemoglobin subunit alpha | HBA1 |
| BRCA1 | Breast cancer type 1 susceptibility protein | BRCA1 |
| 物种编号 | 物种 |
|---|---|
| HUMAN | Homo sapiens |
| RAT | Rat |
| PEA | Garden pea |
SNP ID 命名
命名并不统一,NCBI中对所有提交的snp进行分类考证之后,都会给出一个rs号,也就是参考snp,并且会给出相关的snp信息,包括前后序列、位置信息、分布频率等。一般的命名是:feature ID后面加7-8位数字,如rs12345678或者dbSNP|rs12345678
UCSC ID
其实之前还有ucsc的数据,uc+3位数字+3位字母但是现在已经差不多被放弃
参考:
- https://www.ncbi.nlm.nih.gov/books/NBK143764/
- https://asia.ensembl.org/info/about/index.html
- https://guangchuangyu.github.io/cn/2017/03/bed%E6%96%87%E4%BB%B6/
- 关于HGNC的ID转换https://www.jianshu.com/p/07663121c0d0
- 关于GO:http://fhqdddddd.blog.163.com/blog/static/1869915420128289474633/
- 关于refseq http://liucheng.name/379/
- 关于dbSNP http://blog.sina.com.cn/s/blog_751bd9440102w6rm.html
- 直播基因组67: clinvar数据库
- 重新了解一下clinvar数据库 https://mp.weixin.qq.com/s?src=11×tamp=1539611864&ver=1184&signature=iPRDX8lNVuGrNwemlfTeXXrSxOOly802EBhgYP0DgHtW2WqnxJVQ*GXTsW0mYAxn57EZAnXog29Tg08hit7exmHprwqJuBLt-NoBC8ahcSuFzhl2uNl1Ek2tIfZpCZYP&new=1