251-资源推荐——Rstudio server教你学各种组学分析
刘小泽写于2021.8.6 生信学习从不缺少资源,这不偶然看到一个好的R组学分析学习资源,和你分享
前言
看到生信界的一大活跃人物Sean发了个推文,说有个bioc workshop,点进去看了下确实资源很多,并且还提供免费的Rstudio server来练手

链接在:http://app.orchestra.cancerdatasci.org/1
举个例子
目前有60多个workshop,涵盖了很多组学分析,比如rnaseq、scrna、atac、宏基因组、hic等等,竟然还有中文版本
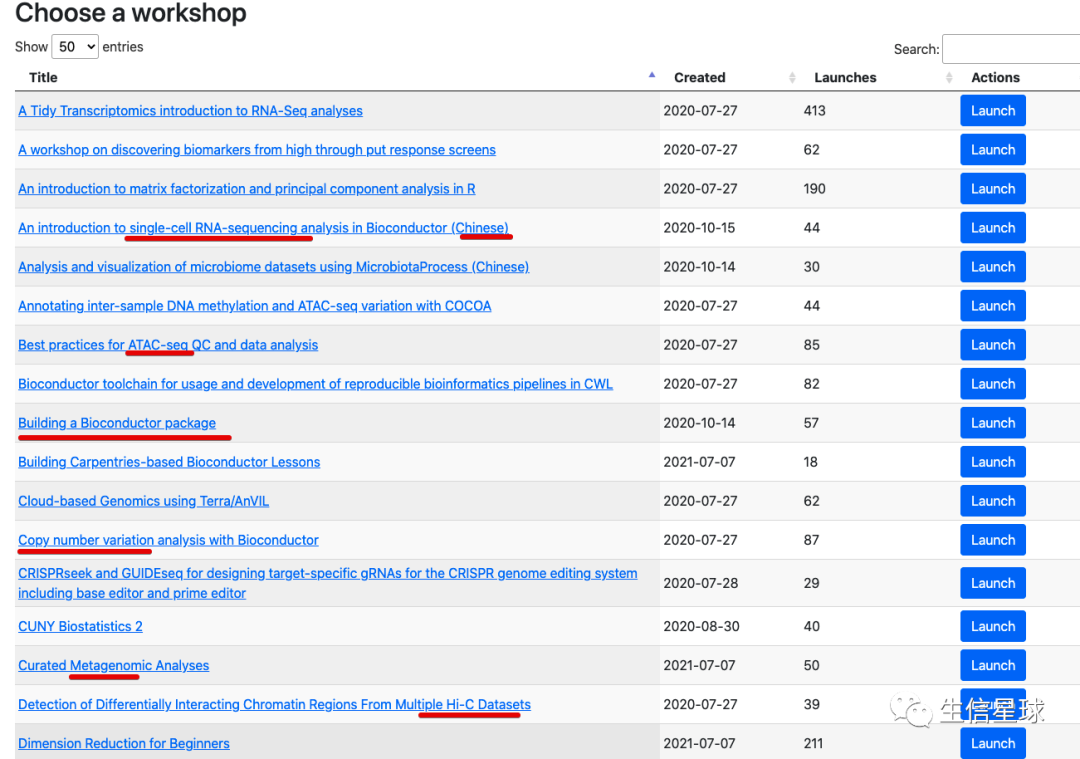
点击右边的launch就会跳转到Rstudio server,不过需要等待一会才能进入
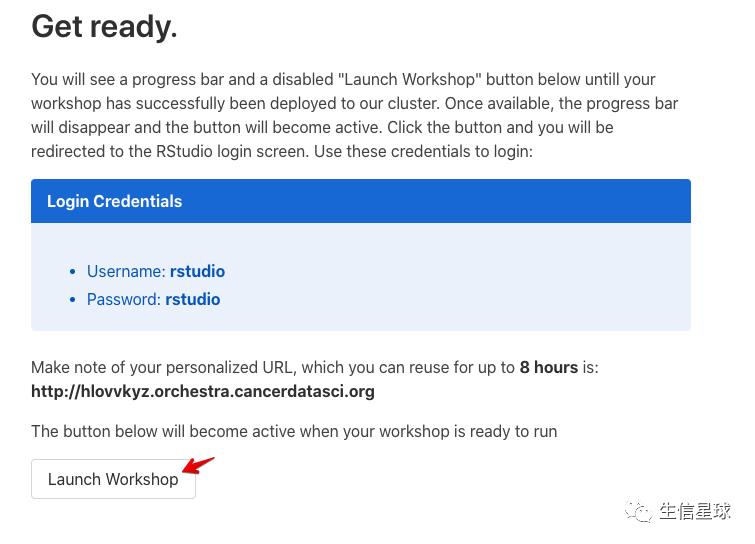
我们就来看一眼这个中文版的scrna分析吧:
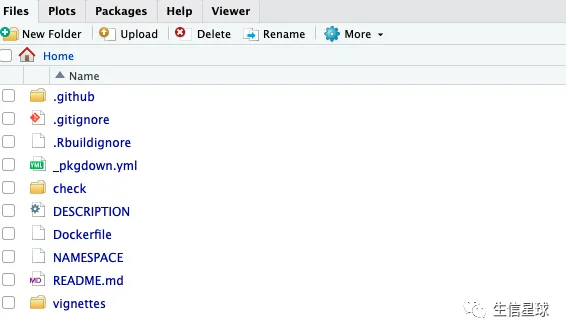
首先看readme
主要包含以下几个点,这些应该都是学员比较关心的问题(需要准备什么、学多久、怎么学、能学到什么):
- Workshop Description
- Pre-requisites
- R / Bioconductor packages used
- Time outline
- Learning goals
- Learning objectives
- Analyze a 10x dataset which contains 4K cells (https://support.10xgenomics.com/single-cell-gene-expression/datasets).
- Run basic steps included in scRNA-seq analysis.
- Learn how to interpret on scRNA-seq analysis generated results.
- Take away some tips and tricks on generating related plots.
然后就是它的操作部分,做成了Rmd文件

基本上使用scater + scran走了一下标准流程的前半部分
- 数据准备(下载10x PBMC4k数据并读取)
- 质量控制
- 数据标准化
- 特征选择 + 降维
- 聚类分析
- 使用
louvain算法进行聚类:buildSNNGraph + igraph::cluster_louvain - 寻找聚类特异表达基因:
findMarkers
- 使用
- 使用
singleR进行细胞类型注释
不得不说,现在cellranger + seurat的组合是最常用的了,但是分析工具的选择也不拘泥于它们,还可以尝试其他的,比如我在之前写的:**
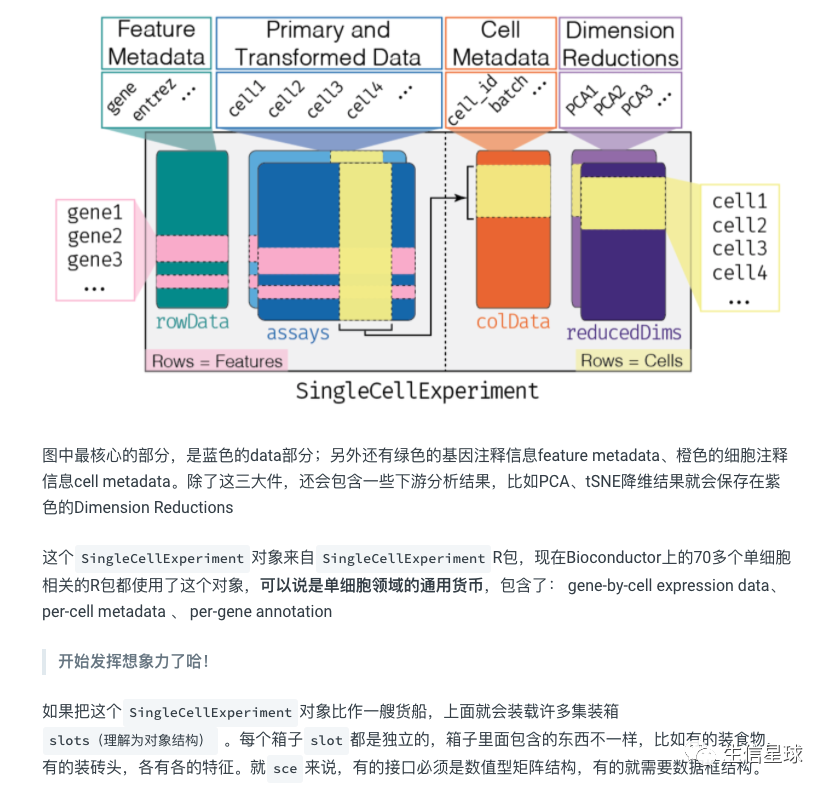
《单细胞交响乐》**就大量使用了scater以及SingleCellExperiment这个数据结构