177-大佬推荐必是精品
刘小泽写于2020.3.19 偶然看到大佬又在推荐精品资源,于是进去看了看,确实写得很走心,和你分享可以详细学习学习 资源就是:Bioinformatics Workbook(https://bioinformaticsworkbook.org/list.html) 里面会提供原始数据供下载练习,并且提供了详细的脚本,应该有几十个超链接,每个打开平均能学1个小时左右
入门-命令行使用
- Unix基础
- 集群HPC使用
- SSH技巧
- Bioawk使用
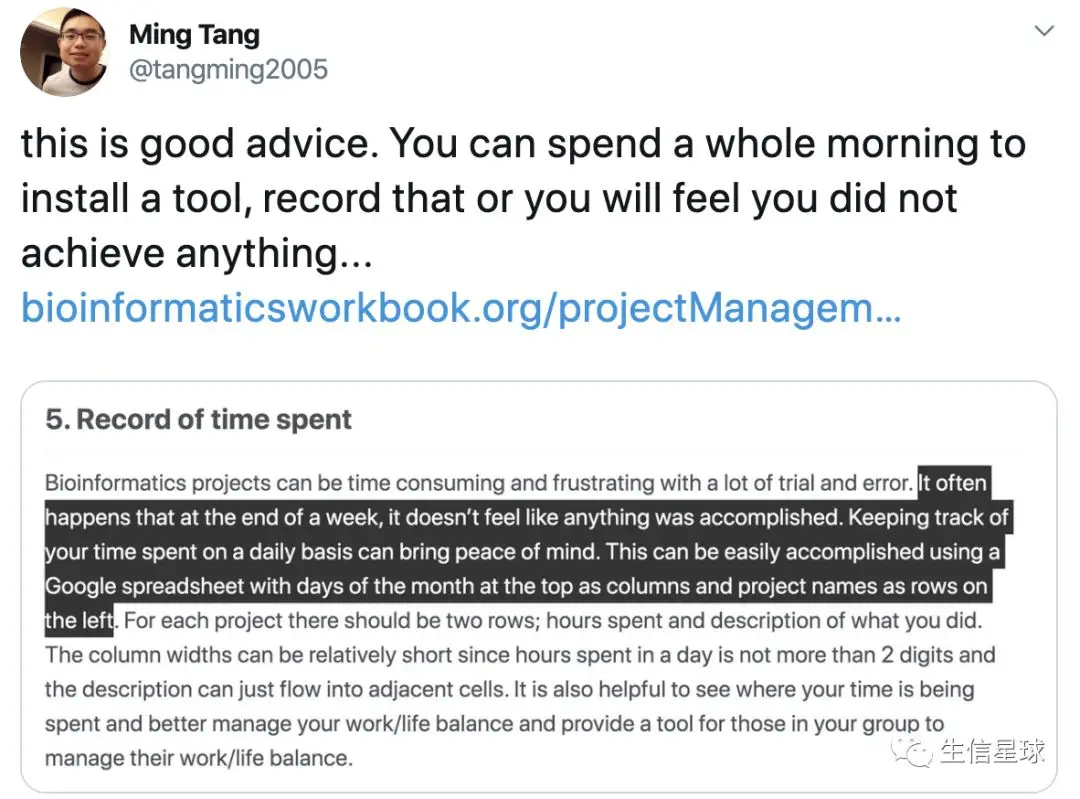
效率-项目管理
- 如何组织服务器数据
- markdown笔记组织
- todolist
BLAST使用
- 示例讲解
- 批量运行
实验设计相关
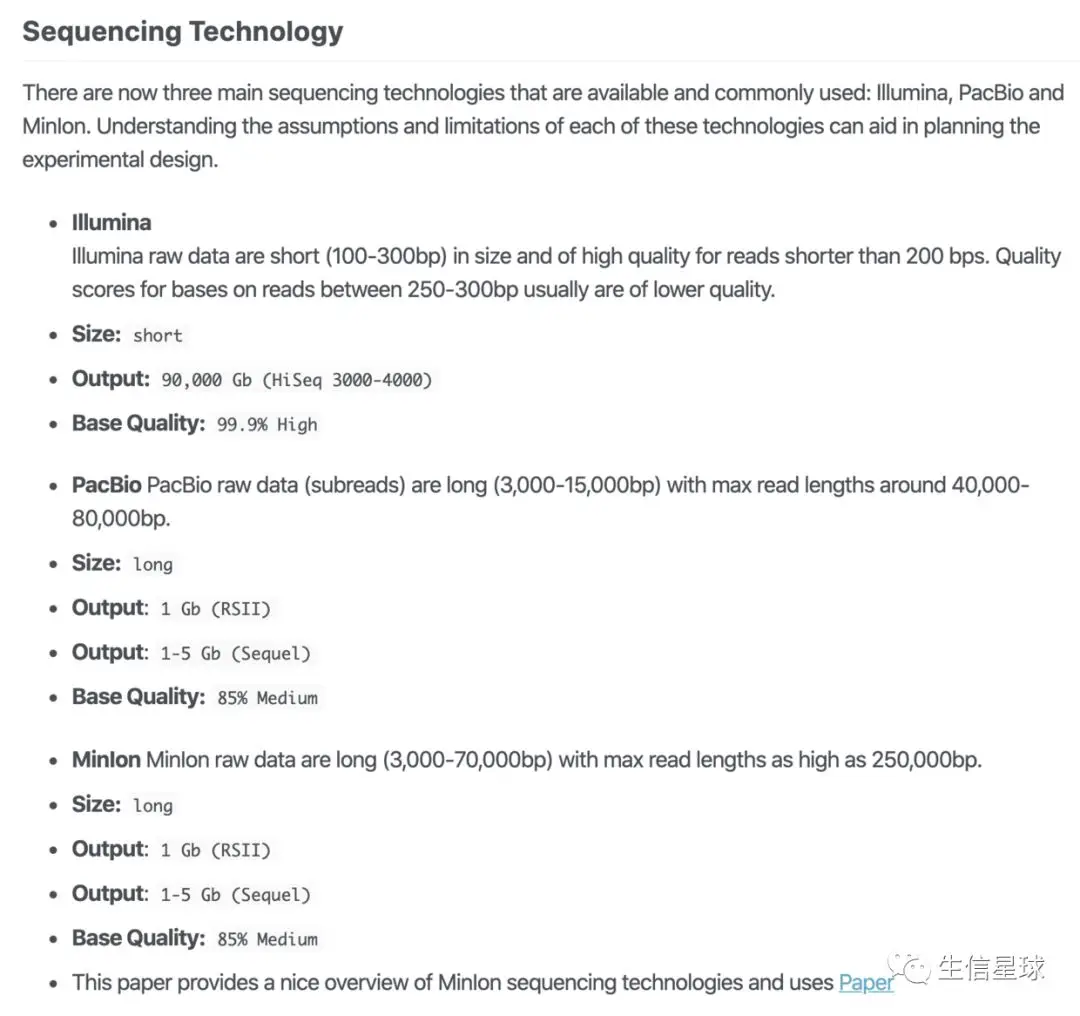
数据获取和处理
- 数据传输
- fasta格式处理
- excel数据处理
- NCBI SRA数据管理
生信格式介绍
- Reads, Contigs, Scaffolds and Chromosome
- 文件格式:FASTA、FASTQ、GFF、GTF、VCF、SAM、BAM
- 质量值
转录组分析
有参
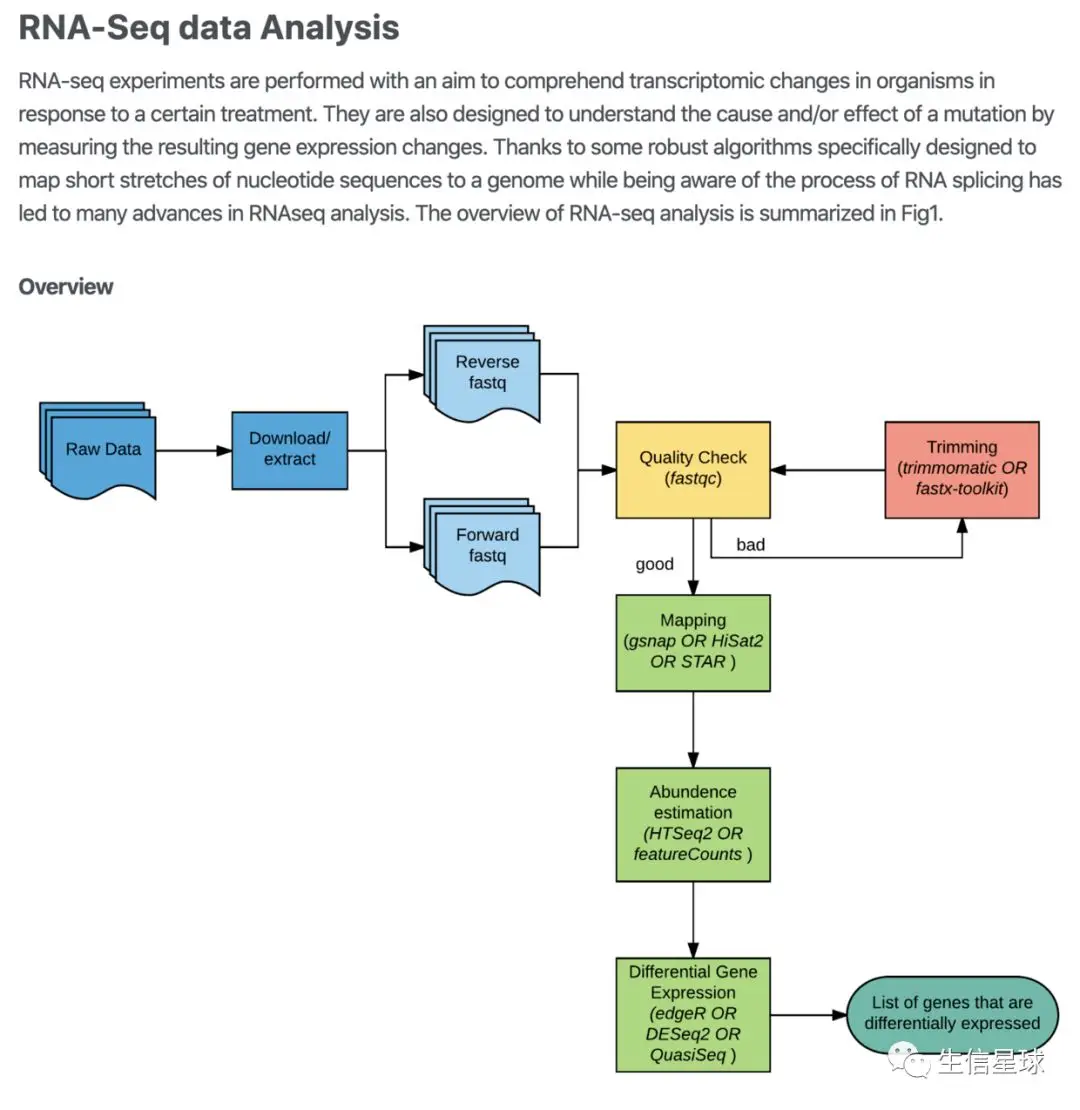
从下载数据开始练习:

无参
差异分析
10X单细胞转录组练习
基因组组装和注释
- 组装多个软件:Canu、SPAdes、 MaSuRCA
- 注释:Maker Gene Prediction、Braker2 Gene Prediction、motif鉴定(MEME and FIMO)
- 组装质量检测:GenomeScope、UniVec、PhiX
- Scaffold组装:Pyscaf、Hi-C scaffolding
- 示例练习:苏云金杆菌数据、拟南芥数据
- 遗传图谱构建
比较基因组学
- 学习Opscan, Iadhore and Circos
- 用R包处理:GeneOverlap 、phylostratr
找变异
- FreeBayes variant calling workflow for DNA-Seq
- GATK Best Practices Workflow for DNA-Seq
- SNP calling for GBS data using Stacks pipeline
- SNP calling for GBS data using Tassel pipeline (GBS.v2)
基因组重复序列鉴定
- Helitron Identification in a Genome Sequence
- DNA Transposon Annotation with Inverted-Repeats Finder
- LTR Retrotransposon Annotation with LTR-Finder
- Repeat Annotation from Next-gen Sequencing Reads Using RepeatExplorer
- De-Novo Repeat Identification and Annotation from Genome Assemblies using RepeatModeler and RepeatMasker
- Tandem Duplication Annotation in a Genome Assembly Using Mummer and RedTandem
数据可视化
- 简单的查看命令
- R语言示例代码演示boxplot、heatmap
- 基因组组装的gaps可视化
- DotPlots for comparing genomes