164-R语言做的GO富集结果和在线工具不一致?真的是这样吗
刘小泽写于2020-2-9 最近被问到一个问题,为什么同样的差异基因用在线工具:http://geneontology.org/ 得到的GO富集分析结果和用R语言得到的不同?为了解决这个问题,我又得学习一下从来不用的在线工具,技多不压身,下面就来看看我的探索过程
模拟使用的基因ID
为了更好地再现整个过程,我会使用相同的基因,均使用Ensemble ID
先来模拟50个差异基因
ENSG00000116299
ENSG00000092621
ENSG00000265972
ENSG00000196208
ENSG00000138434
ENSG00000115461
ENSG00000145911
ENSG00000130707
ENSG00000124942
ENSG00000103257
ENSG00000229807
ENSG00000125148
ENSG00000031698
ENSG00000234741
ENSG00000115935
ENSG00000109321
ENSG00000164171
ENSG00000182199
ENSG00000096696
ENSG00000065911
ENSG00000136068
ENSG00000184575
ENSG00000117983
ENSG00000164690
ENSG00000102024
ENSG00000266714
ENSG00000160180
ENSG00000167757
ENSG00000197043
ENSG00000082175
ENSG00000101144
ENSG00000100889
ENSG00000113494
ENSG00000118513
ENSG00000113719
ENSG00000162981
ENSG00000115468
ENSG00000105974
ENSG00000046604
ENSG00000124225
ENSG00000232677
ENSG00000091527
ENSG00000065361
ENSG00000155850
ENSG00000114405
ENSG00000196405
ENSG00000135473
ENSG00000130396
ENSG00000168003
ENSG00000042062
首先用在线工具
进入http://geneontology.org/,然后把上面的基因ID复制到输入框,然后它会使用panther进行富集分析
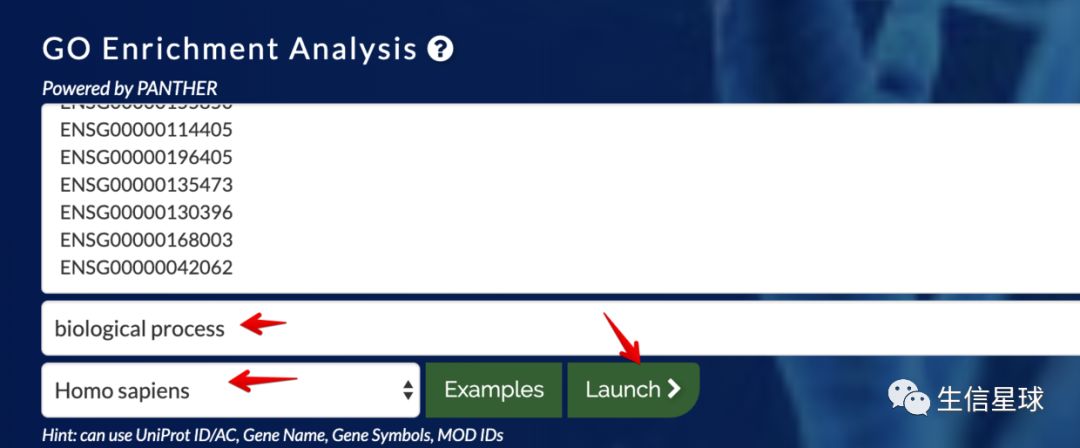
然后结果很快就拿到了,看到结果中有这么几个内容
- 使用FDR进行p值矫正
- 背景基因是20996个,我们提供的50个基因中有3个基因没有对应的GO注释
- 点击table可以下载结果,其中包含的都是矫正后的p值小于0.05的结果(先下载下来,一会再用)

查看这个结果中有多少的GO term
目的就是拿到图中的GO:xxxxxxx
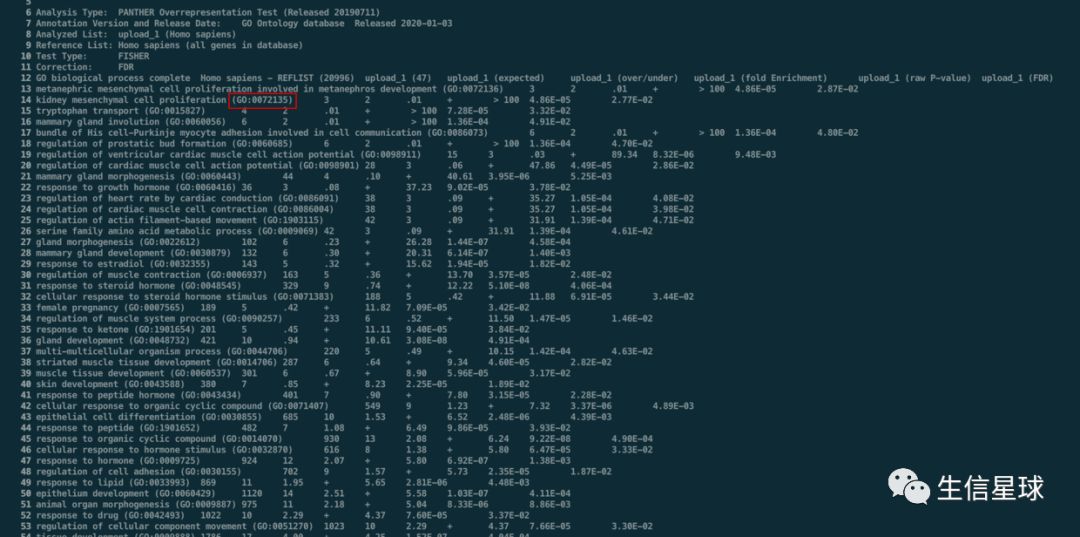
# 能达到目的即可
cat analysis.txt | cut -f1 | grep "GO:" | cut -d "(" -f2 | cut -d ")" -f1 >panther_go.csv
# 结果有49个term

然后使用R语言
suppressMessages(library(org.Hs.eg.db))
suppressMessages(library(clusterProfiler))
# 先将这50个基因保存为gs.csv
gs <- read.csv("gs.csv",header = F)
gs <- gs$V1
go <- enrichGO(gene = gs,
keyType = 'ENSEMBL',
OrgDb = org.Hs.eg.db,
ont = "BP",
pAdjustMethod = "fdr",
pvalueCutoff = 0.05,
qvalueCutoff = 0.05,
readable = TRUE)
go <- as.data.frame(go)
> dim(go)
[1] 137 9
发现有137条结果

看到结果中的背景基因是20610个,我们有7个基因不存在GO注释。可能是由于使用的数据库没有官网新,一个是2019年7月的,一个是2020年1月的

看看二者的区别
# 读取panther结果
test <- read.table('panther_go.csv')
panther_go <- test$V1
> length(panther_go)
[1] 49
# R的结果
r_go <- rownames(go)
# 比较一下
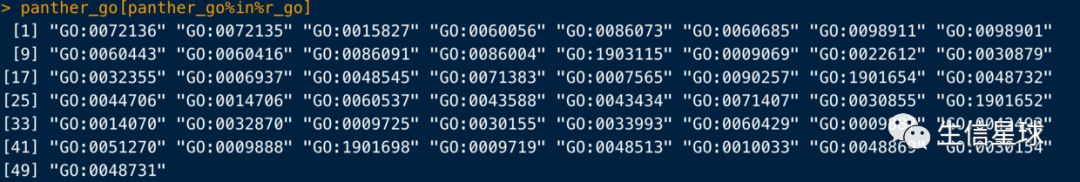
panther_go[panther_go%in%r_go] # 23个
panther_go[!panther_go%in%r_go] # 26个

看到,虽然R得到的结果比panther多,但是依然存在许多不同的GO term
思考 🤔为什么会存在不同的term?
最首先想到的是数据库的差异,但差异应该也不会这么大
那么最直接的检查办法就是挑几个不同的term检查一下
那就先挑选二者结果不一致的第一个term:GO:0072136
放在这个网站检查一下:http://www.informatics.jax.org/vocab/gene_ontology/GO:0072136
【注意链接,举一反三,其他的term也知道怎么查了】

当我看到这个结果的时候,我恍然大明白
是什么导致了panther结果有,而R的结果没有呢?是这个term中包含的基因的数目 如果没有想到什么,那么就应该去看看
enrichGO的帮助文档,其中有两个重要的参数,我们一般会忽略掉,但其实很有意义:minGSSize = 10, maxGSSize = 500
其实之前Y叔回答过这个问题,请看:https://github.com/YuLab-SMU/clusterProfiler/issues/46,就是为了过滤一些特别大的term或者特别小的term,它们会干扰我们的视线(比如特别大的biological process这个term)

并且其中还给出了更好的解决方案DOSE::gsfilter:

# 可以体验一下
data(geneList)
de=names(geneList)[1:100]
x=enrichDO(de)
x
gsfilter(x, min=150, max=170)
为了保证panther的结果,R也有,我将参数范围放大了
这只是为了证明,R分析的结果是靠谱的,不输在线工具。
go_extreme <- enrichGO(gene = gs,
keyType = 'ENSEMBL',
OrgDb = org.Hs.eg.db,
ont = "BP",
pAdjustMethod = "fdr",
pvalueCutoff = 0.05,
qvalueCutoff = 0.05,
readable = TRUE,
minGSSize = 1,
maxGSSize = 30000)
# 其实从上面我们就知道了,通过下面这样就能得到原来的结果
gsfilter(go_extreme, min=10, max=500)
再次进行检验
一个不拉,全包含进来了,并且还比之前panther的49条term多了很多选择。但是多的这些真的有用吗?不一定,但至少不至于没得选
go_extreme <- as.data.frame(go_extreme)
> dim(go_extreme)
[1] 328 9
> panther_go[!panther_go%in%r_go]
character(0)
最后
可能未来还会遇到很多结果比较的需求,尤其是比较在线工具和代码 在实际的科研中,虽然不好说孰优孰劣,但我🤓挺代码