142-如何理解GO富集结果?
刘小泽写于19.10.24 一篇很短的推文,来说明GO的结果
前言
我们使用GO注释的R包做出来的结果一般是长这样:
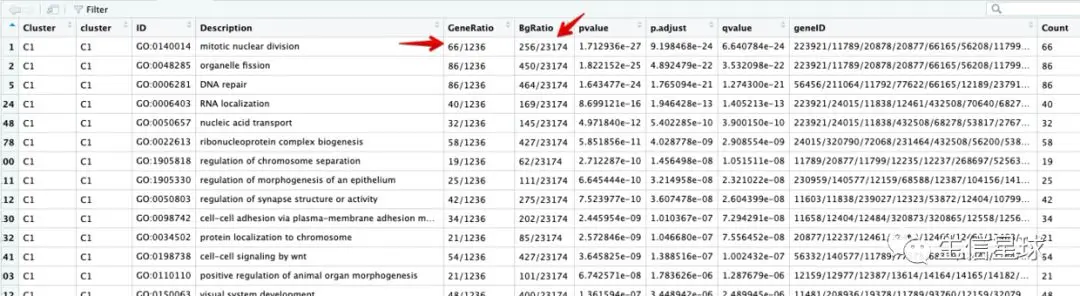
其中重点是这几个数字
看第一行:
- 背景基因是20000多个,其中属于这个GO通路的有256个基因;
- 然后C1这个cluster这里总共得到的差异基因是1236个,其中属于这个GO通路的是66个
那么怎么证明呢?
以第一个GO:0140014为例
首先获得GO:0140014中的基因 =》 256个
因为这是小鼠的注释结果,所以先加载它的物种注释包
library(org.Mm.eg.db)
# 结果有3百多万条GO注释信息
go2gene <- toTable(org.Mm.egGO2ALLEGS)
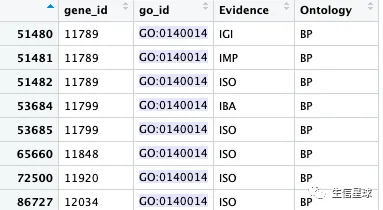
其实看到里面的基因ID存在重复,一个go_id会对应多个同样的gene_id,那么就要去重复,获取unique gene id
uni_gene <- unique(go2gene$gene_id[go2gene$go_id == 'GO:0140014'])
> length(uni_gene)
[1] 256
结果和👆的BgRatio中记录的一致
然后拿到C1的差异基因 =》 1284个
这里的C1指的是第一群细胞中的差异基因(上图中除了这个C1还有C2、C3、C4,表示不同类群的细胞),这个可以不用管,只要知道它是一个分类就可以
# 这一行代码不能运行,只是为了说明上面图中的C1
c1_genes <- list_de_gene_clusters[['C1']]
> length(c1_genes)
[1] 1284
看到这里也是1000多个,不过和上面表格中得到的1236差了几十个基因,这也就说明了存在几十个基因没有GO注释
找Cluster1在GO:0140014中的基因 =》 66个
其实就是找c1_genes和go2gene的交集
overlap_genes <- intersect(c1_genes,uni_gene)
> length(overlap_genes)
[1] 66
这个值就是对应上面GO通路的66个基因