136-看个StateQuest视频学习ChIP-seq
刘小泽写于19.9.11 距离上次学习表观遗传知识过去了大半年(表观遗传初识~ChIP-seq:https://www.jianshu.com/p/30bbc88771d0),之后也没有涉足。但是这一块估计会在我未来的学业中用到,所以还是很有必要拾起来的
视频地址在:https://www.youtube.com/watch?v=nkWGmaYRues&t=226s
染色质、染色体、DNA、组蛋白关系
从前有群细胞,细胞有个核,核里有群染色体,染色体又是特殊时期的染色质,染色质又是由DNA+组蛋白+其它蛋白
根据 百科解释:真核细胞的细胞周期中,大部分时间是以染色质的形态而存在。染色质是间期细胞遗传物质存在的形式,由DNA、组蛋白、非组蛋白及少量RNA 组成的线性复合结构;染色体是指细胞在有丝分裂或减数分裂过程中,由染色质聚缩而成的棒状结构。两者化学组成没有差异,只是包装程度不同。

DNA包裹着组蛋白,形成一个一个的小DNA包裹,然后这个小包裹就可以调控基因表达。至于激活还是抑制基因表达,主要看组蛋白是怎么修饰的。至于包裹的过程这里不探索,只要知道染色质包含了DNA及包裹的组蛋白即可。

用一根线作为模型进行解释
- 因为是线性结构,所以用蓝线代表染色质(但实际上,这根线不是直线,而是像上图一样)
- 褐色箭头表示基因
- 绿色圆圈表示允许转录的组蛋白
- 红色八边形表示抑制转录的组蛋白
- 细胞中会有各种各样的蛋白可以结合到DNA上(图中不同颜色,不同性状)
- 左边黄色的蛋白假设是促进基因转录的
- 右边粉红的蛋白假设是抑制基因的
- 中间绿色的可能有其他未知的功能

那么怎么研究这些蛋白的作用到底是什么?就可以用ChIP-seq
ChIP-seq表示:Chromatin Immunoprecipitation Sequencing 表示染色质免疫共沉淀测序,它的作用就是定位那些DNA结合的蛋白在基因组上的位置
如果我们想要知道绿色蛋白结合的所有区域

首先,用甲醛等试剂将原来结合在DNA上的蛋白固定在DNA上(glue all of the proteins bound to the DNA together with the DNA),但事实上这个操作会把所有蛋白都固定住,不止是我们感兴趣的绿色的蛋白=》这个过程中文翻译是“交联”;
然后,将固定后的DNA切成小片段(大约300bp)
接着,针对我们感兴趣的蛋白,我们加入与它对应的特异性抗体(下图黑色为抗体),然后抗体只和感兴趣蛋白之间结合

然后,回收所有抗体,这样也就把感兴趣蛋白和对应的DNA一起收回来了,并将其他“杂质”去掉
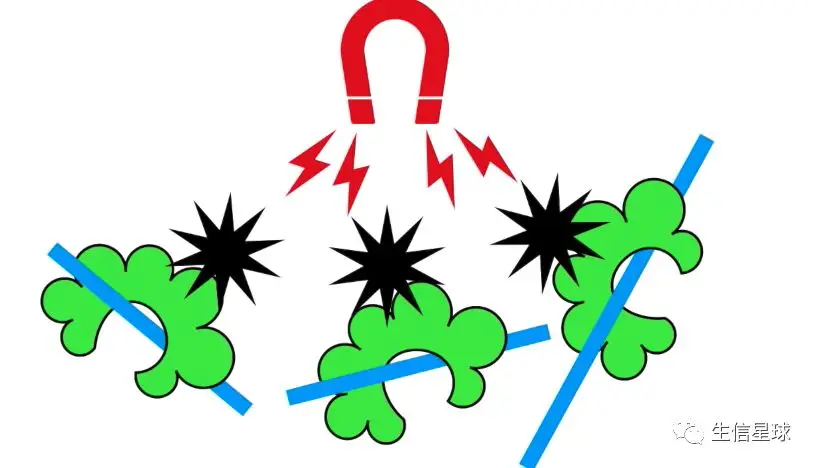
然后,通过加热将蓝色线条(包括DNA和组蛋白)和蛋白分离=》也就是“去交联”过程。接着将蛋白去除,只留下DNA(黑色线条)
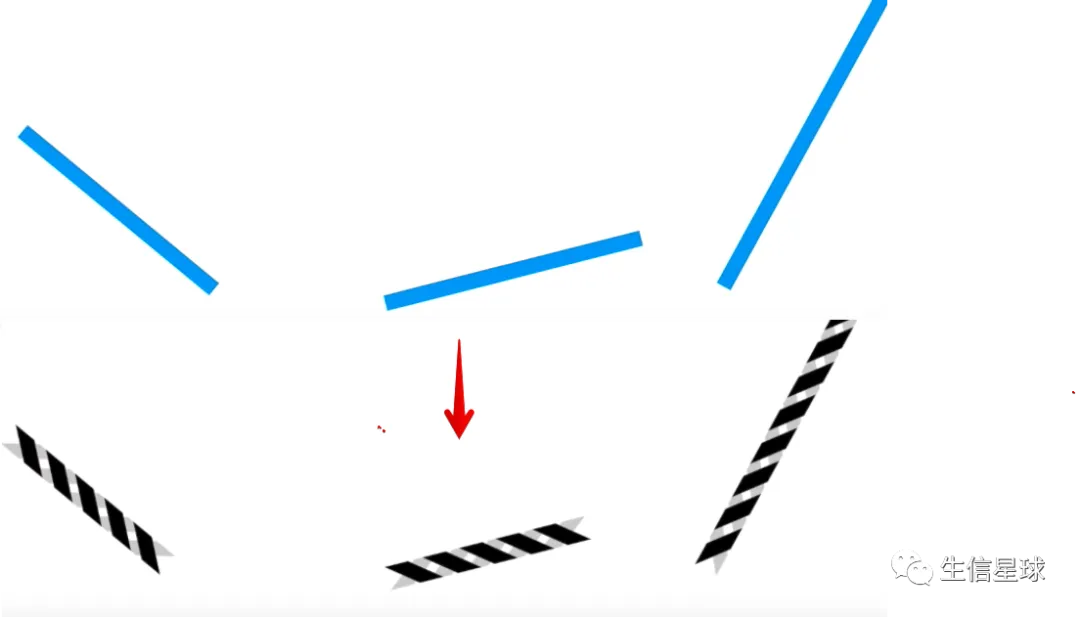
实际操作
以上操作是针对一个细胞中的一条染色质进行解释的,实际实验过程中,一般会对6百万的细胞进行这样的处理,因此我们最后会得到来自大量细胞的大量DNA片段
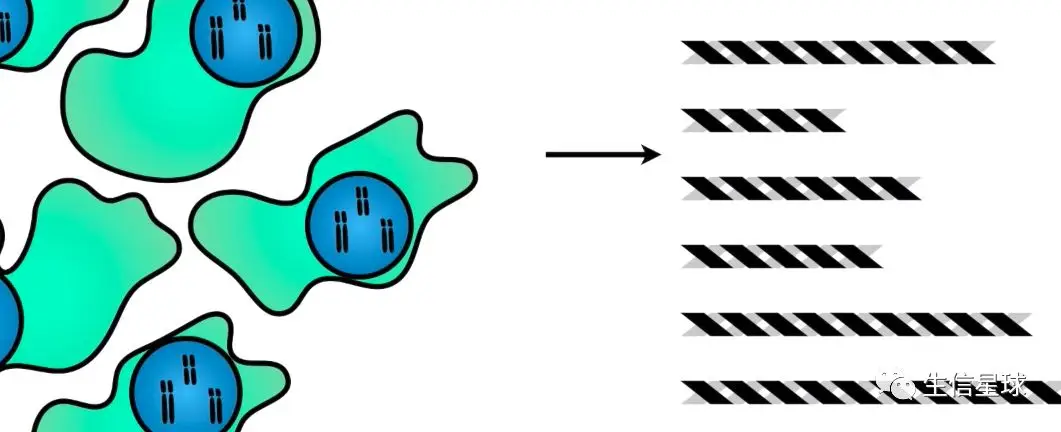
然后这些序列就会经历:
- PCR扩增文库
- 检查文库浓度
- 测序
- 过滤低质量序列
- 比对得到位置信息
例如下图就是比对后的reads对应的染色质位置信息:每个reads来源都不同

最后,会得到所有reads的一系列基因组坐标(一般会有50M到100M的reads)

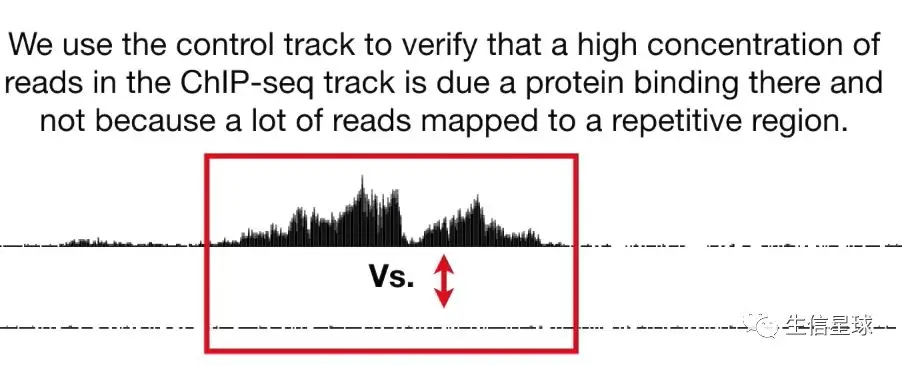
然后利用这些坐标,可以以基因组为背景,构建一个track信息,看看哪个区间内的reads多,由于reads数量多,区间划分细,所以最后有的reads多的地方,就会形成一个峰。下面👇这个图有三部分内容:
- 顶部是基因组中的基因信息,包含了位置、长度、外显子等(mm10)
- 中间是我们利用ChIP-seq的reads创建的一个track,中间两个黑山峰就说明有很多reads比对到了这个区域;而两侧的其他区域比对的reads就很少
- 底部也是一个track,它作为对照。设置对照的原因是考虑到了ChIP-seq实验中,如果染色质被打断成的片段不均匀,就会导致测序reads在基因组上的分布不均匀。对照组一般是从免疫沉淀之前的DNA样本(就是直接利用图3的交联结果,打断,而不使用特异性抗体)中提取一部分DNA,结果会得到各种各样的蛋白对应的DNA序列 ,后续处理和我们样本的ChIP-seq数据一模一样
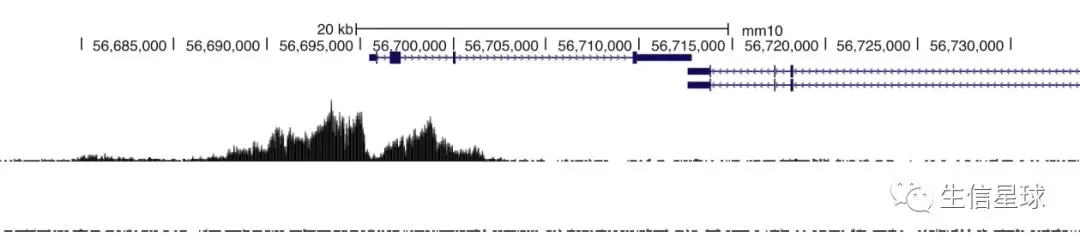
利用对照组,就可以证明:这个区域reads的高占比是由于这里有蛋白结合,而不是因为这个区域是重复序列导致比对很多次
应用
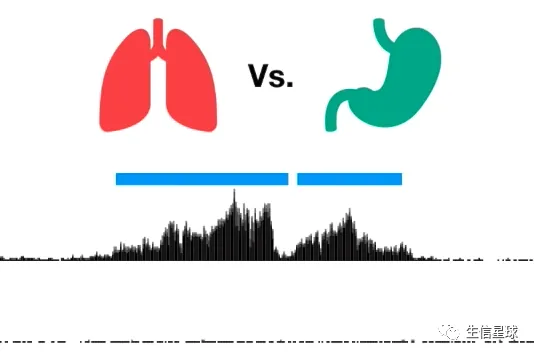
比较同一蛋白在不同组织中的差异,例如:
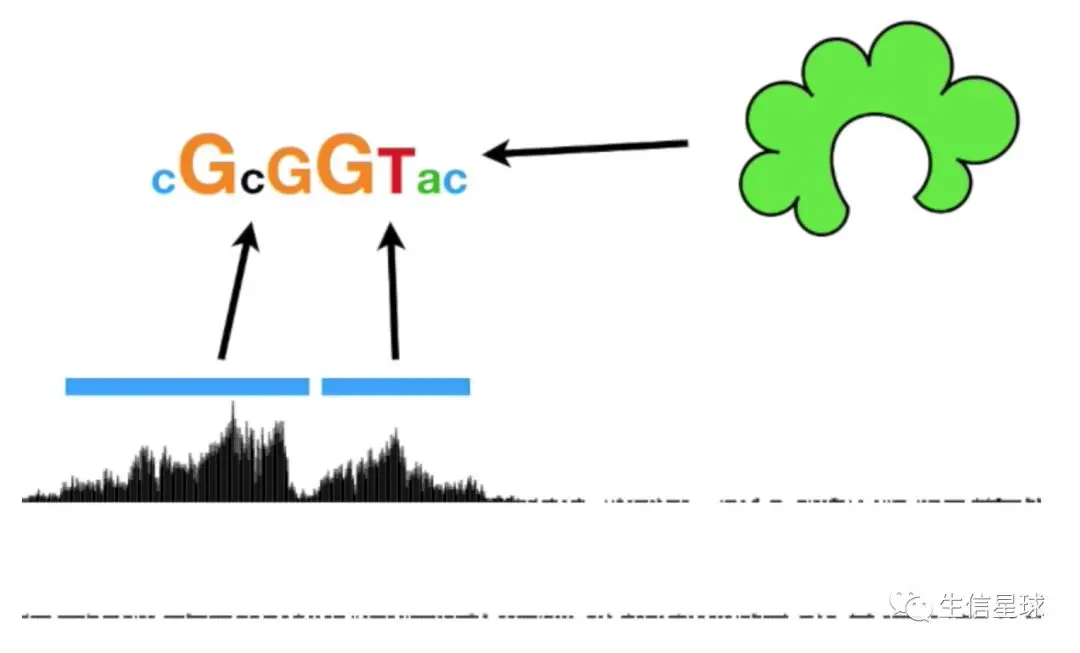
如果不知道某个蛋白结合的序列是什么,可以猜测它是一个在所有peaks都出现的motif,进行motif分析。字母越大,表示在这个蛋白结合序列中出现的频率更高
通过看结合位点所在基因区域,尝试解释结合蛋白的功能,下图看到这个蛋白结合到了基因起始位点,所以推测它可能与基因调控有关
