102-生物变异背景知识~逃不过躲不掉,终究要知道
刘小泽写于19.4.10 学习癌症知识不可避免要遇到一个问题,什么是somatic mutation?什么是germline?和SNV、CNV又有什么关系?SNP和SNV是一回事吗?还有非同义突变、错义突变、无义突变又是怎么一回事?这些生物变异相关的背景知识逃不过躲不掉,终究要知道
癌症种类有 至少200种癌症以及更多的亚型,癌症与正常组织区别之一就是癌症的基因组一些位点发生了改变,而有的改变驱动了肿瘤细胞的生长,而有的改变无关痛痒,我们就是想看看那些比较核心的基因变化。
首先来看somatic和germline mutation
看三个问题:
**1 肿瘤样本和正常样本哪里不同?**这个问题的解决就是"call mutation"过程(可以用
GATK、varscan、freebayes)2 得到的突变都是患者本身发生的吗? 这个问题的解决就是"call somatic mutation”。这个因为在癌症研究中,初步得到的变异是患者包含从父母那里得到的变异(germline变异或胚系突变),还有一部分是我们真正关心的患者本身因为环境因素等造成的变异(somatic变异或体细胞突变)。实际操作中一般都是得到肿瘤与正常配对组织一起进行WGS或WES测序,这样做的目的就是过滤掉从亲本得到的germline 变异,获得真正属于研究对象的somatic变异,也就是我们想看的(可以用
Varscan、SomaticSniper、MuTect2、MuSE、Strelka可以检测,其中前四个是TCGA使用的)。3 得到体细胞突变后,其中有许多突变并不是直接导致癌症发生的,那么真正致癌基因怎么获得? 得到的体细胞突变又可分为两种类型:驱动突变和乘客突变(driver & passenger)。这个问题的解决就是"driver gene mutation”,就是寻找那些驱动基因。2018年有一篇Cell文献 Comprehensive Characterization of Cancer Driver Genes and Mutations,他们使用26种工具分析了TGCA MC3项目(Multi-Center Mutation Calling in Multiple Cancers project)体细胞突变集,最终确定了299种癌症基因,3400个假定的错义驱动突变基因,实验验证60~85%的预测结果是可能的驱动基因
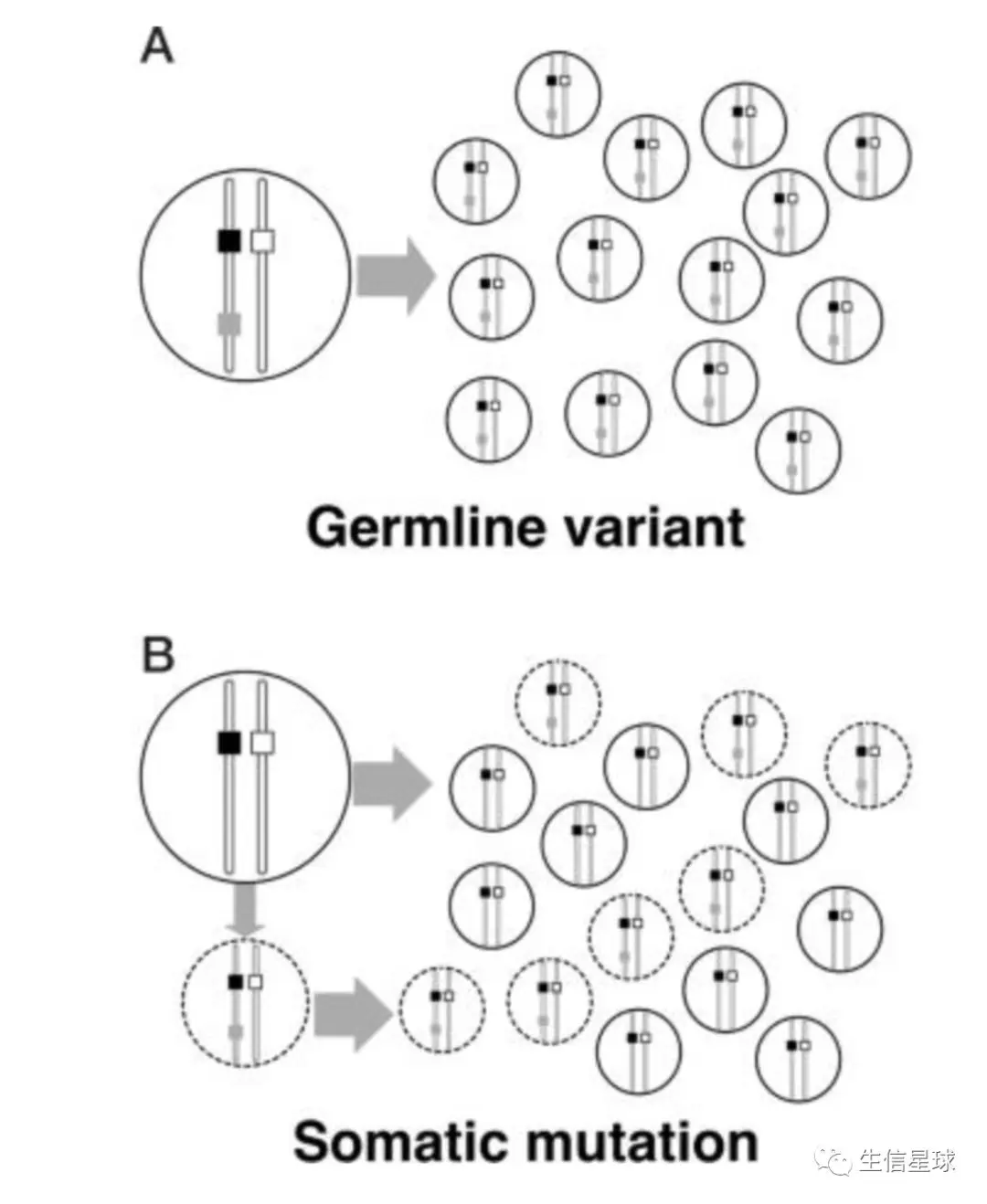
第一次接触这两个词是从GATK软件中,开始并不清楚什么意思,只能找图片帮助理解,于是看到这一张:

先上英文解释:
- Somatic mutations – occur in a single body cell and cannot be inherited (only tissues derived from mutated cell are affected)。体细胞突变基因简称SMGs
- Germline mutations – occur in gametes and can be passed onto offspring (every cell in the entire organism will be affected)
关于基因变异Genetic Variation的分类:
单核苷酸畸变Single Nucleotide Aberrations
- SNPs:single nucleotide polymorphism单核苷酸多态性=》mutations shared amongst a population
- SNVs:single nucleotide variation单核苷酸变异=》private mutations
短插入缺失:Short Insertions or Deletions (indels):插入或缺失小于50bp的small indel(也有文献是25bp)
拷贝数变异:Copy Number Variations (CNVs):片段扩增或缺失
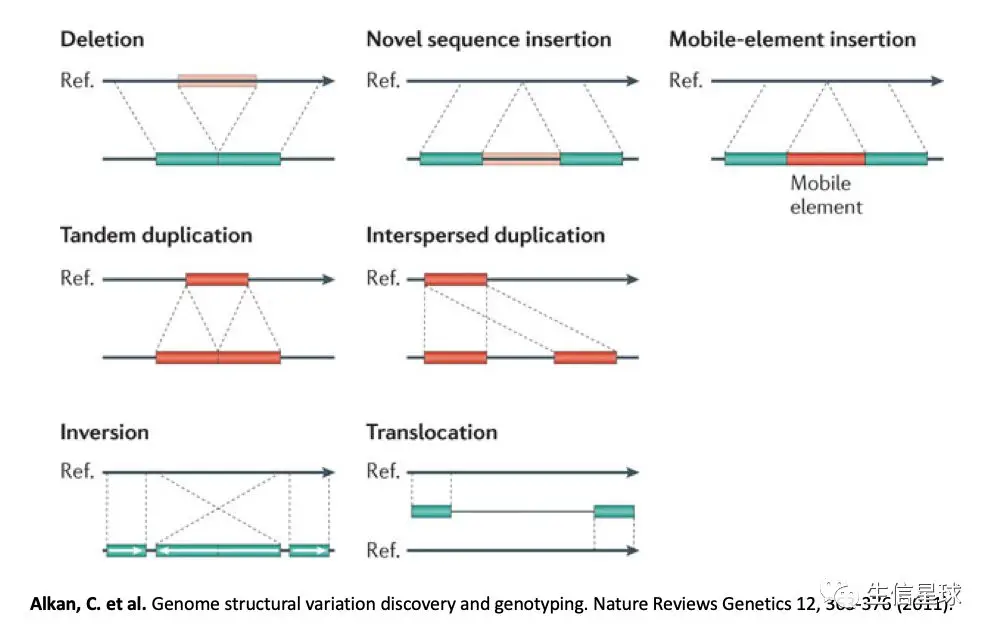
结构变异:Larger Structural Variations (SVs):基因组水平上大片段插入、缺失、倒置、易位等,其中易位包括染色体内部迁移和染色体间的迁移
点突变:SNV与SNP
实体肿瘤中95%的突变为单个碱基的替换
这两个经常分不清楚,但共同点在于都是针对单个碱基;
不同点在于:
- SNV:就是个体存在的变异位点,是一种低频突变,在群体中没有被证实
- SNP:有一个"群体"的概念包含其中,就是说一个群体中的任何个体都存在变异可能,而且是一种高频突变。信息储存在dbSNP (http://www.ncbi.nlm.nih.gov/snp)
上面看到一个关键词:“低频、高频”,这也体现出二者的区别主要是在多态性上。这里的频率指的是 minor allele frequency(MAF),即次要等位基因频率。再细致一点就是一个种群中出现频率排名第二的等位基因(allele),例如三个等位基因GG、GC、CC,出现频率分别为0.50,0.48,0.02,那么MAF就是0.48
知道了频率的定义,那么这个数值怎么界定呢?这个就依据不同文献了,有的文献定为5%,有的定为1%,即位点突变频率低于1%是低频,反之为高频
因此可以看到,SNP更倾向于反应一个群体的突变频率=》多态性;SNV检测的是针对个体,它也是应用在somatic变异检测中的指标之一
更多参考:https://www.quora.com/Genomics-What-is-the-difference-between-an-SNP-and-an-SNV
https://www.researchgate.net/post/What_is_the_difference_between_a_SNP_and_a_mutation
点突变又分为=》同义突变|错义突变|无义突变
- 同义突变(silent mutation, same-sense or synonymous mutation):密码子是有简并性的,因此单个碱基的替换可能只改变了mRNA上特定的密码子,但是不改变翻译时氨基酸的正常编码
- 非同义突变又包括错义突变和无义突变
- 错义突变(missense mutation or non-synonymous mutation):单个核苷酸改变导致一个密码子编码一个不同的氨基酸
(可以利用基于蛋白质序列的保守程度的
SIFT和基于序列、结构特性的Polyphen2、只用于错义点突变的MutationAssessor、整合前三者的OncodriveFM等进行功能预测http://college.gcbi.com.cn/archives/1653) - 无义突变(non-sense mutation):一个密码子变为终止密码子引起多肽链合成提前终止,产生的蛋白大都失去了活性或丧失了正常的功能。
- 错义突变(missense mutation or non-synonymous mutation):单个核苷酸改变导致一个密码子编码一个不同的氨基酸
(可以利用基于蛋白质序列的保守程度的
CNV 拷贝数变异
基因组发生重排而导致的,长度1k以上,可以按照CNV对否致病分为:致病性CNV、非致病性CNV和不明临床意义CNV。
CNV突变率较高,大约是DNA点突变的100-10000倍
目前全基因组范围内研究CNV的方法主要有:基于芯片的比较基因组杂交技术(Array CGH, aCGH)、SNP分型芯片、NGS染色体异常检测
Database of genomic variants, DGV数据库 收录了已报道的CNVs
参考: 基因组拷贝数变异及其突变机理与人类疾病; CNV的临床意义
小结:
绝大多数癌症的突变是体细胞突变(大约90%的癌基因显示有体细胞突变,20%的显示有胚系突变,而10%的显示共有体细胞和胚系突变 A census of human cancer genes);
体细胞变异主要联合分析配对肿瘤和正常样本序列,然后看:SNV、CNV、InDel
最后是人类基因变异的一些参考数据库
The 1000 Genomes Project
- http://www.1000genomes.org/
- SNPs and structural variants from 2500 individuals from about 25 populations
HapMap
- http://hapmap.ncbi.nlm.nih.gov/
- identify and catalog genetic similarities and differences
dbSNP
- http://www.ncbi.nlm.nih.gov/snp/
- Database of SNPs and multiple small-scale variations
COSMIC
- http://www.sanger.ac.uk/genetics/CGP/cosmic/
- Catalog of Somatic Mutations in Cancer
TCGA
- http://cancergenome.nih.gov/
- The Cancer Genome Atlas researchers are mapping the genetic changes in 33 cancer types
ClinVar
- ·http://www.ncbi.nlm.nih.gov/clinvar/
- aggregates information about sequence variation and its relationship to human health